学生アルバイトKさんのLigandScout関連論文の紹介記事です.LigandScoutの活用事例としてご参考になれば幸いです.
(以下,論文紹介記事)
本記事では、次の論文を、LigandScoutの利用を中心に紹介します。
In silico and in vitro identification of candidate SIRT1 activators from Indonesian medicinal plants compounds database
(Azminah Azminah, Linda Erlinac, Maksum Radji, Abdul Mun’ia, Rezi Riadhi Syahdi, Arry Yanuar, Computational Biology and Chemistry Volume 83, December 2019, 107096)
https://doi.org/10.1016/j.compbiolchem.2019.107096
サーチュイン(Sirtuin)は、クラスIIIヒストン脱アセチル化酵素であり、NAD+依存性脱アセチル化酵素群の総称です。その中の一つサーチュイン1(SIRT1)の活性化合物は、2型糖尿病、老化プロセス等に対して保護的な役割を果たしている可能性が示唆されています。著者らは、ファーマコフォア手法に基づくバーチャルスクリーニングにより、インドネシアの薬草に含まれる1377個の化合物の中からサーチュイン活性化合物となり得る4個の化合物を見出しました。
本論文における解析手順は下図の通りです(Graphical abstract)。
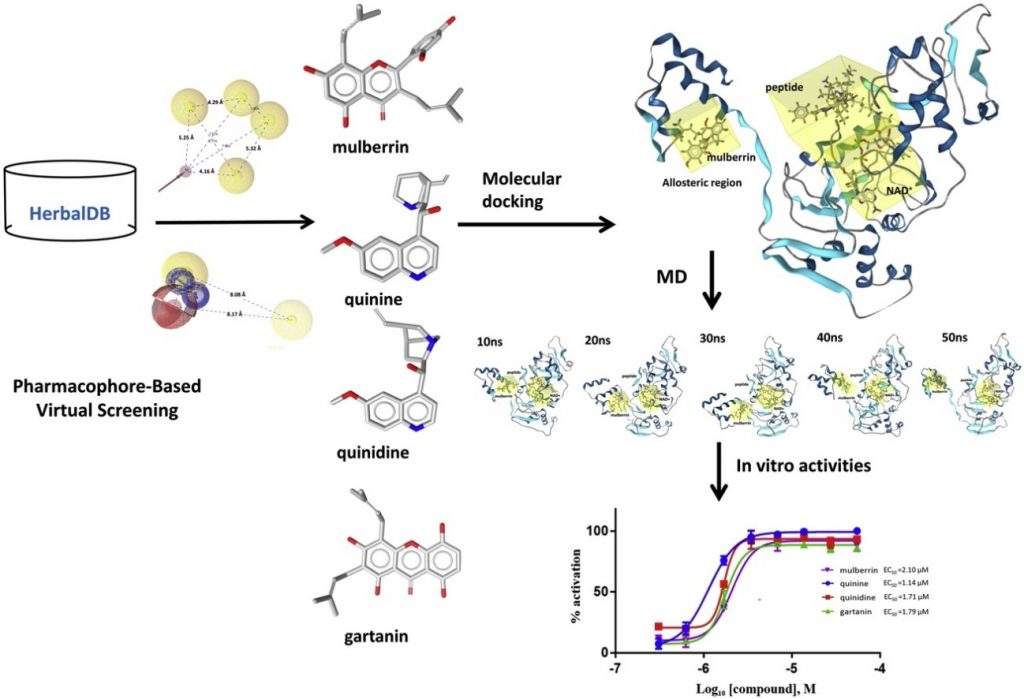
Graphical abstract
化合物探索
候補化合物の探索には、Pharmacophore-Based Virtual Screeningが採用されており、LigandScout 4.2が使用されています。LigandScoutでは、構造ベースとリガンドベースの2つの手法からファーマコフォアモデルの作成が可能です。構造ベースモデルは、ヒトSIRT1のPDBファイル(PDBID:4ZZJ)のアロスティック結合部位からファーマコフォアを作成しています (Fig. 2) 。リガンドベースモデルは、既知のサーチュイン活性化合物を、12個のトレーニングセットと25個のテストセットに分けて、前者を用いて10個のファーマコフォアモデルを構築しています。その後、構造ベースとリガンドベース、両ファーマコフォアモデルのバリデーションが行われています。LigandScoutでは、活性化合物及びデコイ化合物を用いてスクリーニングを行うことで、ファーマコフォアモデルの性能指標となるパラメータ(sensitivity, specificity, accuracy, receiver operating characteristic (ROC), areas under the curves (AUC:1,5,10,100%), enrichment factors (EF:1,5,10,100%))を求めることができます。筆者らは、テストセットとした既知のサーチュイン活性化合物25個とDirectory of Useful Decoys : Enhanced (http://dude.docking.org/generate)を用いて作成したデコイ化合物1392個を用いてファーマコフォアモデルの評価とリファインメントを行っています。この結果、構造ベースは条件(AUC1,5,10,100%全て0.5より大、かつEF1,5,10,100%全て1より大)を満たしたため、そのまま採用、またリガンドベースモデルでは最も性能のよかったModel 1を採用しています (Fig. 3) 。
その後、構造ベースとリガンドベース2つのファーマコフォアをクエリとして1377個の薬草化合物データベースを対象としたバーチャルスクリーニングが実施されています。構造ベースファーマコフォアモデルからは10個、リガンドベースファーマコフォアモデルからは18個の化合物がヒット化合物として得られました。
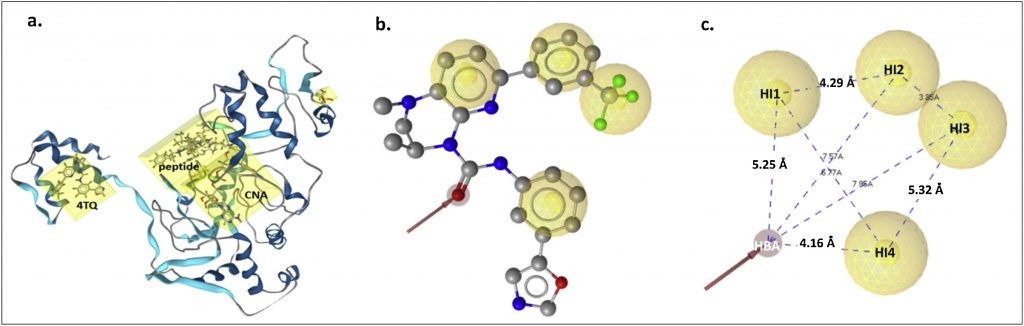
Fig. 2. LigandScoutにより作成された構造ベースファーマコフォア (a) SIRT1 (b) 3Dファーマコフォア (c) 共結晶化された4TQリガンドのファーマコフォア要素(HI1/HI2/HI3/HI4:疎水性相互作用,HBA:水素結合受容体)と距離
Table 1. リガンドベースファーマコフォアモデル(Model 1-10)の曲線下面積(AUC)と濃縮係数(EF)、Sensitivity、Specificity、Accuracy
| Model | AUC100% | EF1% | Sensitivity | Specificity | Accuracy% |
| 1 | 0.88 | 12.1 | 0.84 | 0.91 | 91.1 |
| 2 | 0.86 | 8.1 | 0.80 | 0.91 | 90.8 |
| 3 | 0.84 | 12.1 | 0.92 | 0.63 | 63 |
| 4 | 0.90 | 16.2 | 0.96 | 0.60 | 61 |
| 5 | 0.76 | 0.0 | 0.96 | 0.47 | 48 |
| 6 | 0.88 | 16.2 | 0.96 | 0.58 | 58 |
| 7 | 0.90 | 16.2 | 1.00 | 0.44 | 45 |
| 8 | 0.78 | 20.2 | 0.92 | 0.62 | 60 |
| 9 | 0.79 | 16.2 | 0.92 | 0.62 | 65 |
| 10 | 0.92 | 12.1 | 1.00 | 0.62 | 46 |
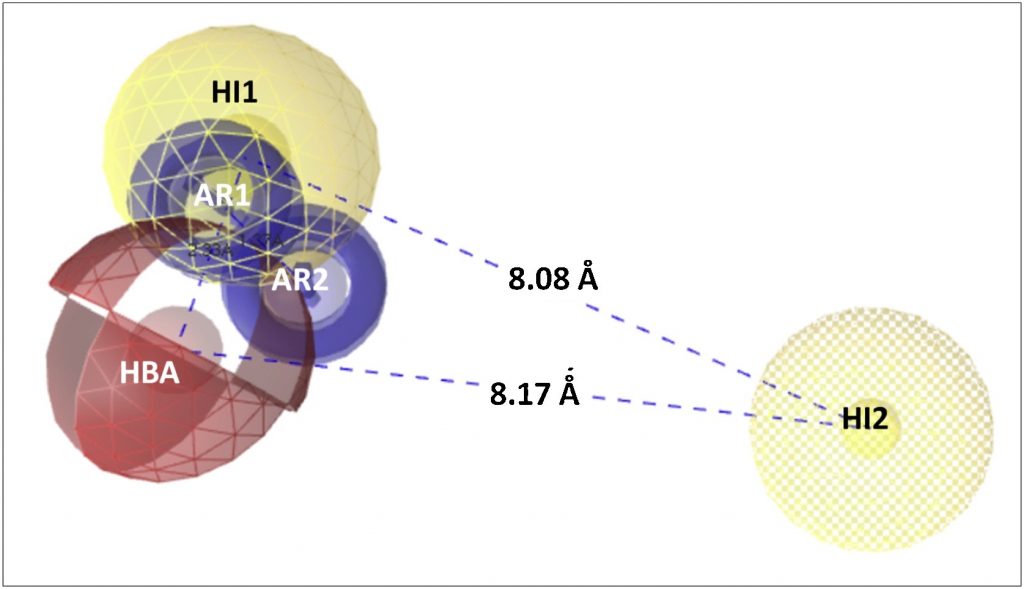
Fig. 3. リガンドベースファーマコフォア Model 1. 構成要素(HI1/HI2:疎水性相互作用,AR1/AR2:芳香環, HBA:水素結合受容体)と距離
ヒット化合物の絞り込み
Pharmacophore-Based Virtual Screeningにより得られた28個のヒット化合物に、リピンスキーのルール・オブ・ファイブ(molecular weight<500, HBA<10, HBD<5, clogP <5)を適用し、薬剤候補として適した化合物を絞り込みました。結果として、構造ベースファーマコフォアモデルによるヒット化合物からは1個(Mulberrin)、リガンドベースファーマコフォアモデルによるヒット化合物からは3個(Quinine、Quinidine、Gartanin)、計4個の化合物が絞り込まれています。
ヒット化合物の評価
続いて、理論計算と実験の双方から、絞り込んだ4個の候補化合物のサーチュイン活性化合物としての適性が評価されています。
理論計算では、SIRT1とのドッキング計算及びMD計算が行われています。ドッキング計算にはAutoDock4Zn(http://autodock.scripps.edu/resources/autodockzn-forcefield)が利用されており、PDB:4ZZJのリガンドである4TQとスクリーニングで得られた4個の候補化合物とSIRT1とのドッキングポーズ及び相互作用が比較されています(Fig. 4)。Mulberrin、Quinine、Quinidine、Gartaninのドッキングスコアは、それぞれ-4.89、-4.71、-4.98、-5.67 kcal/molでした。また比較のため、サーチュイン活性化合物の一つであるResveratrolとSIRT阻害剤のNicotinamideもドッキング計算されており、各スコアは、-4.29、-4.63 kcal/molでした。これらの化合物と結合部位周辺のアミノ酸残基との相互作用がFig. 5に示されています。
MD計算には、Amber(https://ambermd.org/)が利用されており、ドッキングと同様、4TQと4つの候補化合物について、RMSDとRMSFそれぞれの値、また、MM-GB(PB)SA法によってΔG[kcal/mol]が求められています(Fig. Extra 1)。
in vitro試験では、SIRTの相対的な酵素活性を精製酵素より測定可能な発光アッセイシステムSIRT-Glo™ Assaysが用いられており、4つの候補化合物のEC50値を求めています(Fig. 8)。
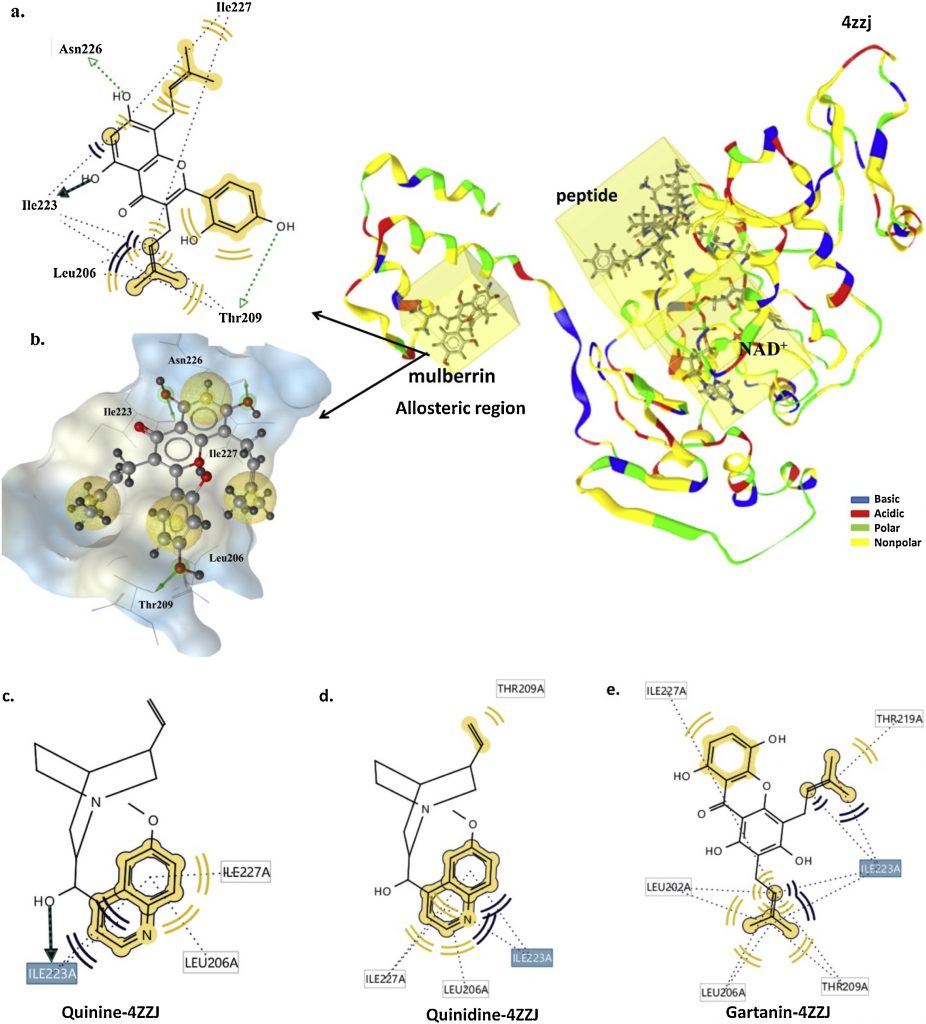
Fig. 4. ドッキング結果に基づく相互作用(いずれもLigandScoutにより作図)(a)4ZZJとMulberrinの2D図 (b)Murberrinの3Dサーフェス図(親水性:青色、疎水性:灰色) (c)Qunine (d)Quinidne (e)Gartanin各2D図
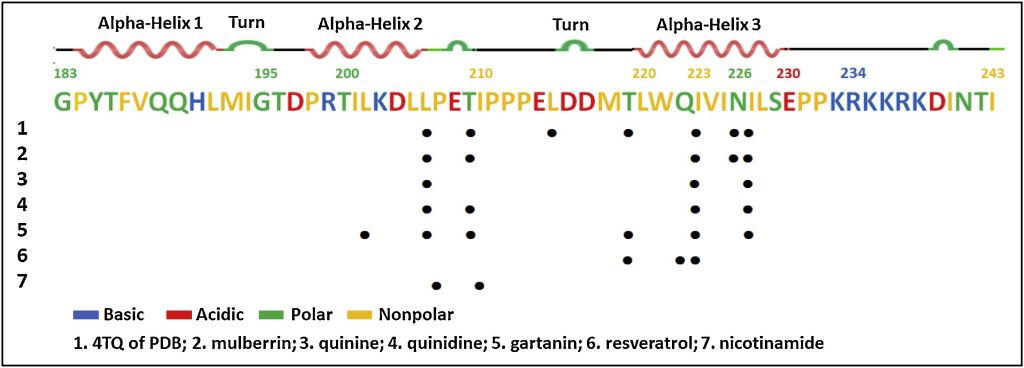
Fig. 5. (1) 共結晶構造(PDB:4ZZJ)における4TQリガンド、並びに(2)Mulberrin、 (3)Quinine、(4)Quinidine、(5)Gartanin、(6)Resveratrol、(7)Nicotinamideの各ドッキング結果に基づくSIRT1アロステリックサイトのアミノ酸残基との相互作用
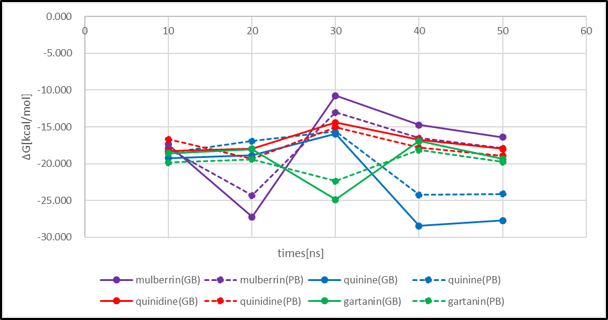
Fig. Extra 1. MM-GB(PB)SA法による結合自由エネルギーΔG変化(論文Table 2に基づきグラフ化)
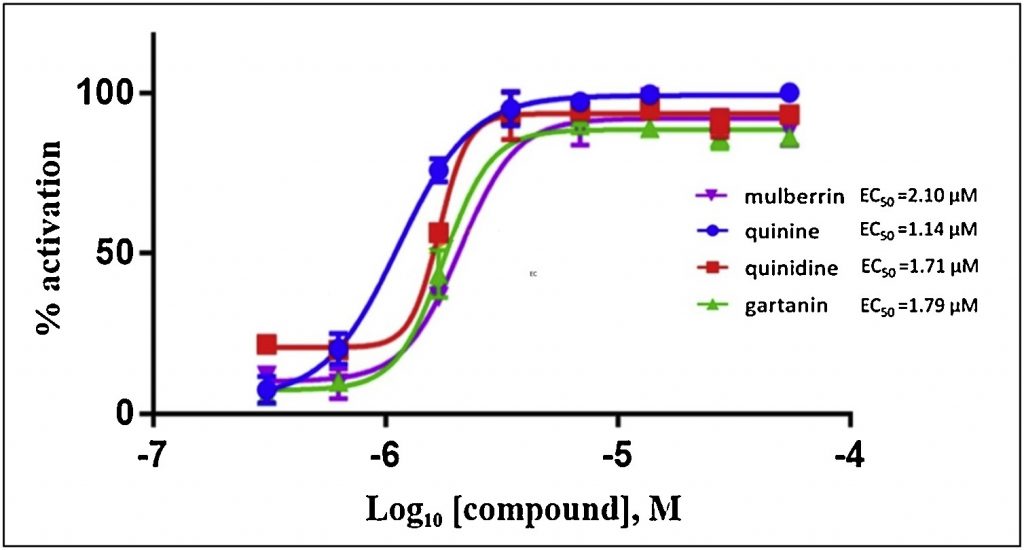
Fig. 8. Mulberrin, Quinine, Quinidine, Gartaninの活性プロファイル(SIRT-Glo™)
結論
インドネシアの薬草化合物1377個を対象としたファーマコフォアに基づくバーチャルスクリーニングにより、4個の候補化合物Mulberrin、Quinine、Quinidine、Gartaninが選出されました(Fig. Extra 2)。in vitroアッセイの結果、それぞれのEC50値は2.1 µM、1.14 µM、1.71 µM、1.79 µMであり、いずれもサーチュイン活性化合物として適していることが示されています。また、ドッキング解析により疎水性相互作用に重要とみられるIle223とIle227が提示され、MM/PB(SB)SA計算により予測された結合自由エネルギーはin vitroアッセイの結果と良い相関を示しています。
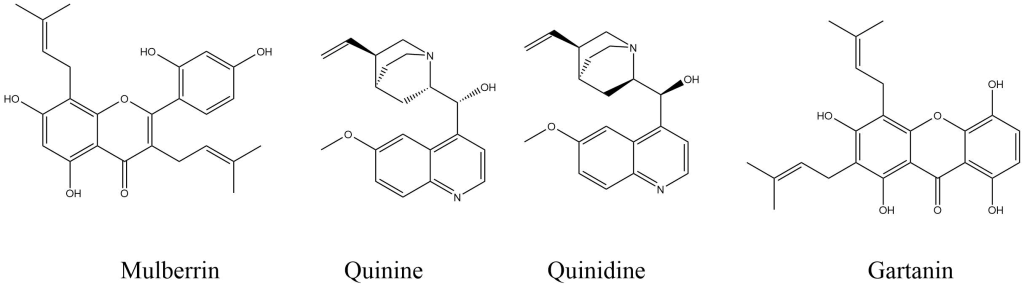
Fig. Extra 2. 見出されたサーチュイン活性化合物
Category: LigandScout