前回に引き続き部分構造検索ネタでいきます。今回はもう少し実用的なプログラムを紹介します。それは医薬品として望ましくない部分構造を持つ化合物をライブラリから排除するプログラムです。日本において購入できる低分子化合物は1000万以上あるのですが、その中には医薬品として問題がありそうな(毒性が懸念される)構造をもつものが沢山あります。大抵は反応性が高く、有機合成に利用する反応試薬などがほとんどですが、インシリコスクリーニングを行う際はそれらを予め排除する必要があります。
部分構造フィルター
創薬のスクリーニングにおいて望ましくない部分構造のリストとして最も有名なのは下図のものではないでしょうか。これはRishtonの論文で紹介されています。これだけでなく他にも排除すべき部分構造があるかもしれませんが、とりあえず下図の構造を排除する簡単なプログラムが”filter.cpp“です。リンクを右クリックしてリンク先を保存して下さい。コンパイルについては以前からほとんど同じコマンドなので、そろそろ書くのが面倒になってきましたが以下のように行います。
$ g++ -std=c++11 filter.cpp -I/home/username/app/include/openbabel-2.0 -L/home/username/app/lib -lopenbabel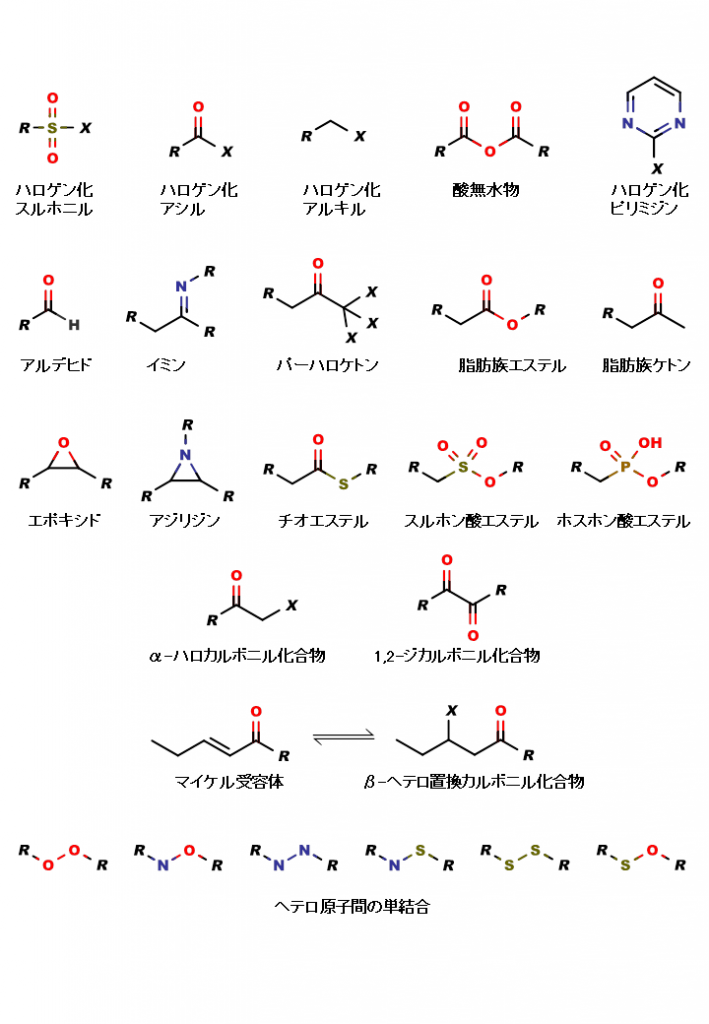
使い方は以下のように入力のSDファイル”input.sdf”と出力のSDファイル”output.sdf”を引数に指定してコマンドを実行します。上記の構造を排除した後の化合物構造が”output.sdf”に出力されます。
$ ./a.out input.sdf output.sdfプログラムを少し解説
このプログラムの本体はFilterクラスです。以下のコードは、このクラスの部分だけ抜粋して示します。Filterクラスのコンストラクタで排除すべき部分構造の初期化と追加をひたすら繰り返しています。追加先はOBSmartsPatternのベクターとなっています。そして化合物のチェックはCheckというメソッドで行っております。該当する部分構造が1個でもマッチした場合はfalseを返して、部分構造が1個もマッチしない場合はtrueを返すようにしています。
このクラスは難しい書き方にはなっていないと思います。独自の部分構造を追加したい場合は、sm.Init(“独自パターン”); sms.push_back(sm);と追記して下さい。問題なのはSMARTSパターンの作り方です。これは試行錯誤しないとなかなかうまく作れません。かなり作りこまないと排除したく無い構造を排除してしまうことになるでしょう。例えば鎖状のエステルを排除したいがラクトンは排除したくない場合はSMARTSの中に環構造ではないという条件を入れなければなりません。
13 class Filter {
14 vector<OBSmartsPattern> sms;
15
16 public:
17 Filter() {
18 OBSmartsPattern sm;
19
20 sm.Init("[F,Cl,Br,I]S(=O)(=O)*"); // ハロゲン化スルホニル
21 sms.push_back(sm);
22
23 sm.Init("[F,Cl,Br,I]C(=O)*"); // ハロゲン化アシル
24 sms.push_back(sm);
25
26 sm.Init("[F,Cl,Br,I][CX4H2]*"); // ハロゲン化アルキル
27 sms.push_back(sm);
28
29 sm.Init("*C(=O)!@-O!@-C(=O)*"); // 酸無水物
30 sms.push_back(sm);
31
32 sm.Init("[F,Cl,Br,I]c1[nH0]ccc[nH0]1"); // ハロゲン化ピリミジン
33 sms.push_back(sm);
34
35 sm.Init("[CX3H1](=O)[#6]"); // アルデヒド
36 sms.push_back(sm);
37
38 sm.Init("*C(C*)=[NX2]*"); // イミン
39 sms.push_back(sm);
40
41 sm.Init("[F,Cl,Br,I]C([F,Cl,Br,I])([F,Cl,Br,I])C(=O)C*"); // パーハロケトン
42 sms.push_back(sm);
43
44 sm.Init("[#6]O!@-C(=O)[#6]"); // 脂肪族エステル
45 sms.push_back(sm);
46
47 sm.Init("*C!@-C(=O)[CX4H3]"); // 脂肪族ケトン
48 sms.push_back(sm);
49
50 sm.Init("O1CC1"); // エポキシド
51 sms.push_back(sm);
52
53 sm.Init("N1CC1"); // アジリジン
54 sms.push_back(sm);
55
56 sm.Init("[#6]S!@-C(=O)[#6]"); // チオエステル
57 sms.push_back(sm);
58
59 sm.Init("[#6]O!@-S(=O)(=O)[#6]"); // スルホン酸エステル
60 sms.push_back(sm);
61
62 sm.Init("[#6]O!@-P(=O)(-[OH])[#6]"); // ホスホン酸エステル
63 sms.push_back(sm);
64
65 sm.Init("[F,Cl,Br,I][CX4H2]C(=O)*"); // α-ハロカルボニル化合物
66 sms.push_back(sm);
67
68 sm.Init("O=C(*)!@-C(=O)*"); // 1,2-ジカルボニル化合物
69 sms.push_back(sm);
70
71 sm.Init("*C[CH]!@=[CH]!@-C(*)=O"); // マイケル受容体
72 sms.push_back(sm);
73
74 sm.Init("*C[CH]([F,Cl,Br,I])!@-[CH2]!@-C(*)=O"); // β-ヘテロ置換カルボニル化合物
75 sms.push_back(sm);
76
77 sm.Init("*O!@-O*"); // O-O結合
78 sms.push_back(sm);
79
80 sm.Init("*N!@-O*"); // N-O結合
81 sms.push_back(sm);
82
83 sm.Init("*N!@-N*"); // N-結合
84 sms.push_back(sm);
85
86 sm.Init("*S!@-N*"); // S-N結合
87 sms.push_back(sm);
88
89 sm.Init("*S!@-S*"); // S-S結合
90 sms.push_back(sm);
91
92 sm.Init("*S!@-O*"); // S-O結合
93 sms.push_back(sm);
94 }
95
96 bool Check(OBMol& mol) {
97 for (auto ii = sms.begin(); ii != sms.end(); ii++) {
98 if (ii->Match(mol)) return false;
99 }
100 return true;
101 }
102 };宿題
本来ならSMARTSのリストを外部ファイルとして作成して、プログラム実行時に読み込ませるような仕様にすべきです。その方が色々と応用が効くし、プログラム自体もスッキリします。今回その様にしなかったのは単純に面倒だったからです。余力がある方は是非このプログラムをSMARTSリストを読み込ませる様に改良してみて下さい。
Category: OpenBabel, プログラミング関連