弊社の提供するin silicoスクリーニングサービスの一つであるCGBVS (Chemical Genomics-Based Virtual Screening)法の新しい応用について紹介します。ある化合物に対してCGBVS法で様々なタンパク質に対するスコアを計算すると、その化合物の特徴ベクトルとして扱うことができます。そしてその特長ベクトルで化合物間のsimilarityを計算することで創薬関連の色々な解析に応用できます我々はこれをCGBSP (Chemical Genomics-Based Similarity Profiling)と呼んでいます。
CGBVSとは?
CGBVS法は機械学習に基づくin silicoスクリーニング法です。in silico創薬において機械学習による手法は、特にタンパク質の立体構造を利用できない場合に使われることが多く、ligand basedの方法として分類されています。もちろんタンパク質の立体構造に基づく方法と組み合わせることもあります。CGBVS法はタンパク質の立体構造を利用しないという意味ではligand-basedの方法なのですが、他のligand-basedの方法と異なる点は、化合物の情報だけでなくタンパク質の情報(アミノ酸配列)も利用するという点です。具体的には化合物-タンパク質の組み合わせが活性を持つかどうかを機械学習するということを行っています。そして、組み合わせを学習データの単位にするメリットとして、複数のターゲットタンパク質に対する同時活性評価が可能になるという点です。あと副効用として私が感じることとして、組み合わせにすることにより学習データがかさ増しされて精度向上に寄与しているとも考えています。
実際のCGBVS法では化合物記述子ベクトルとタンパク質記述子ベクトルのそれぞれを計算して組み合わせたもの(テンソル積)を計算に用います。化合物記述には”alvaDesc”を利用して、タンパク記述子には”PROFEAT2016″などを利用しています。このようにして作成された相互作用ベクトルをSVM (Support Vector Machine)により機械学習して学習モデルが作成されます。学習モデルがタンパク質毎に分かれてなく単一なので、タンパク質間で単純に比較可能なスコアを算出することができます。このような特徴から学習モデルで計算できるタンパク質全部に対して入力化合物のスコアを計算することにより、化合物をプロファイリングできます。このように計算されたプロファイルをから選択性を確認することも可能となります。
CGBVSプロファイリング
弊社のソフトウェア製品であるCzeekSを使えば、コマンド一つで入力した化合物のプロファイルを簡単に計算することができます。CzeekSにはいくつかのモデルが予め作成されていて、GPCR、キナーゼ、イオンチャンネル、核内受容体、プロテアーゼ、トランスポーター、CYP、PPIの8種類があります。これらはChEMBLdbの化合物-タンパク質の相互作用情報を機械学習して作成されております。計算可能なタンパク質数は合計1271個となっています。
CGBVSプロファイリングの例として、アンジオテンシンII受容体拮抗薬のValsartanのGPCRプロファイルを計算してみました。結果は下図のようになります。CGBVSのスコアは0~1までの値を取り、1に近いほど当該タンパク質に作用する可能性が高いと予測されます。このプロファイル図を見るとValsartanには4個のスコア0.8を超えるピークが見られます。左から主ターゲットであるアンジオテンシンII受容体1及び2とGLP1RとPTGDR2 (Prostaglandin D2 Receptor 2)となります。CGBVSでは、これら4個のGPCRに対してValsartan何らかの活性があると予測されます。(アゴニスト若しくはアンタゴニストなど)
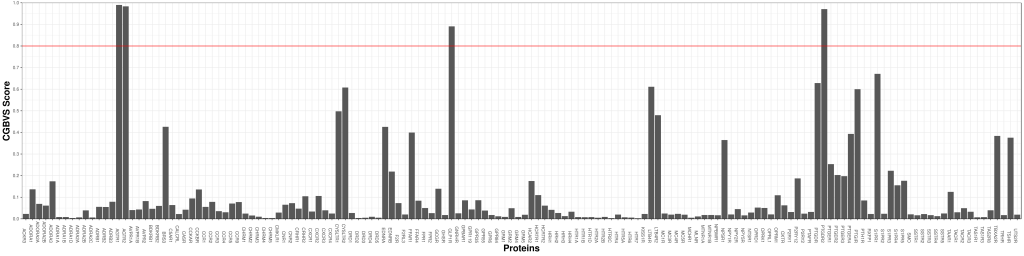
CGBVSプロファイルの類似性
先ほどValsartanのCGBVSプロファイルを計算しましたが、同じアンジオテンシンII受容体拮抗薬のTelmisartanも計算してみました。以下にプロファイル図を示します。
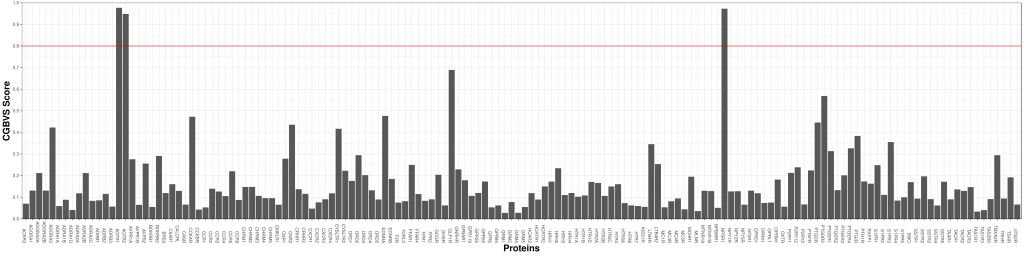
一見した感じでは、どの程度似ているかわかりませんが、これら2化合物のCGBVSプロファイルを特長ベクトルとして扱いsimilarityを計算することができます。2つのベクトル間のsimilarityの計算方法はいくつかあるのですが、我々は単純にcosine similarityを採用しております。一般的にin silico創薬の分野では、単にsimilarityとはfingerprintによるTanimoto係数のことを指すので、我々はCGBVSプロファイルによるsimilarityのことをCGBSPスコアと呼んでいます。この例でのValsartanとTelmisartanとの間のCGBSPスコアは0.816823となりました。一方fingerprint(ECFP4)によるTanimoto係数は0.303226でした。二つのsimilarityは単純比較はできませんが(ランダムサンプリングしたときのsimilarityの平均値を差し引くなどして基準をそろえる必要がある)、明らかにCGBSPスコアの方が良い値となります。このように構造式の比較ではsimilarityが小さくても、CGBVSのsimilarityであれば高い値となります。
今度はCGBSPスコアが小さい例を示しましょう。以下の図はヒスタミンH1受容体拮抗薬の一つであるChlorpheniramineのプロファイル図になります。
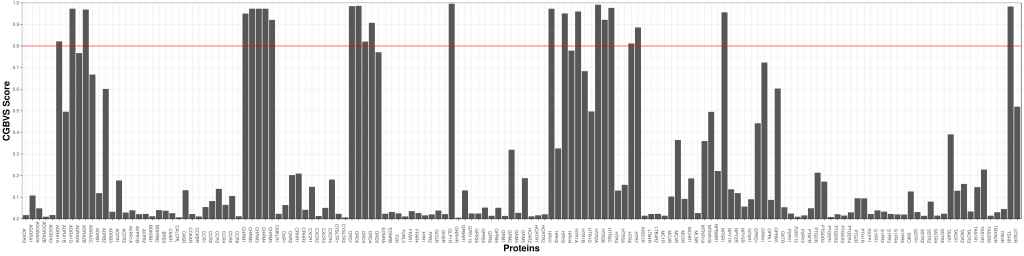
ValsartanとChlorpheniramineとのCGBSPスコアを計算すると0.212261でした。そしてfingerprintによるTanimoto係数は0.261538でした。これはTanimoto係数の方が高いという結果でした。つまり、異なるターゲットの薬とはsimilarityが低いという結果がCGBSPでも得られるということです。
おわりに
ValsartanとTelmisartanのどちらも同一の受容体をターゲットとする薬です。多少構造式が異なっていても両者間のCGBSPスコアが高くなるのはある意味では当たり前なのですが、このような性質があることから現在我々はCGBSPの計算をin silicoスクリーニングに応用できないか検討している最中です。世の中には様々なin silicoスクリーニング方法があるので、今回紹介したアンジオテンシンII受容体のようなターゲットに対して、敢えてCGBSPを適用する意味は無いかと思います。しかしながら創薬研究の状況は千差万別であって、特にタンパク質の立体構造はおろか、何をターゲットとしているかもはっきりしない場合に、活性化合物だけ幾つか存在するという状況でCGBSPが使える場合があると考えています。良い結果が得られたら、何らかの形でご報告できればと思います。
Category: CGBVS/CzeekS, DRAGON/alvaDesc