今回は前編に引き続きOpenBabelを利用した類似構造検索(Similarity Search)についてです。後編ではプログラムを解説します。
プログラム解説
C++プログラムSimSearch.cppのソースコードは以下の通りです。
1 #include <fstream>
2 #include <iostream>
3 #include <iomanip>
4 #include <string>
5
6 #include <openbabel/mol.h>
7 #include <openbabel/obconversion.h>
8 #include <openbabel/fingerprint.h>
9
10 using namespace std;
11 using namespace OpenBabel;
12
13 int main(int argc, char* argv[]) {
14 OBConversion conv;
15 conv.SetInFormat("SMI");
16
17 // フィンガープリントの設定(ECFP4)
18 OBFingerprint* pFP = OBFingerprint::FindFingerprint("ECFP4");
19 // OBFingerprint* pFP = OBFingerprint::FindFingerprint("MACCS");
20
21 // 参照分子の読み込み
22 ifstream ifs1("Fexofenadine.smi");
23 OBMol ref;
24 conv.Read(&ref, &ifs1);
25 ifs1.close();
26
27 // 参照分子のフィンガープリントを取得
28 vector<unsigned int> fp_ref;
29 pFP->GetFingerprint(&ref, fp_ref);
30
31 // テスト分子の読み込み開始
32 ifstream ifs2("test19.smi");
33 vector<unsigned int> fp_mol;
34 OBMol mol;
35 while (conv.Read(&mol, &ifs2)) {
36 // テスト分子のフィンガープリントを取得して谷本係数を計算
37 pFP->GetFingerprint(&mol, fp_mol);
38 double Tc = OBFingerprint::Tanimoto(fp_ref, fp_mol);
39 cout << mol.GetTitle() << "\t" << Tc << endl;
40 // フィンガープリントをクリア
41 fp_mol.clear();
42 }
43 ifs2.close();
44
45 return 0;
46 }先ず8行目でフィンガープリント用のヘッダーファイルをincludeしています。そして18行目で使用するフィンガープリントを設定しています。このプログラムでは”ECFP4″ですが、他にも色々利用できるみたいです。利用可能なフィンガープリントの一覧は”obabel”コマンドで表示できます。
$ obabel -L fingerprints
ECFP0 Extended-Connectivity Fingerprints (ECFPs)
ECFP10 Extended-Connectivity Fingerprints (ECFPs)
ECFP2 Extended-Connectivity Fingerprints (ECFPs)
ECFP4 Extended-Connectivity Fingerprints (ECFPs)
ECFP6 Extended-Connectivity Fingerprints (ECFPs)
ECFP8 Extended-Connectivity Fingerprints (ECFPs)
FP2 Indexes linear fragments up to 7 atoms.
FP3 SMARTS patterns specified in the file patterns.txt
FP4 SMARTS patterns specified in the file SMARTS_InteLigand.txt
MACCS SMARTS patterns specified in the file MACCS.txtフィンガープリントの計算は28、29行目と37行目で実行しています。それぞれ参照化合物とライブラリ化合物についてです。参照化合物のフィンガープリントは最初に一度計算すれば良いので、ループの外側で行っています。フィンガープリントの情報を入れる変数の型は”vector”です。unsigned intのサイズは4バイトなので、32ビットということになります。一方、OpenBabelで計算されるECFP4のデフォルトのビット長は4096です。したがって、このベクターのサイズは128(=4096/32)ということになります。
Similarityを計算する2つの構造のフィンガープリントが計算出来たら、38行目の”OBFingerprint::Tanimoto”という関数で谷本係数が計算できます。このプログラムでは参照化合物のフィンガープリントは”fp_ref”という変数に入れて、ライブラリ化合物の方は”fp_mol”という変数に入れています。谷本係数以外のSimilarity metricがあるのかソースを確認してみましたが、どうやら谷本しか無いみたいです。
RDKitとの比較
OpenBabelで計算されるSimilarityが、他のソフトウェアでの計算値とどの程度の差があるか確認してみました。比較対象としてはやはりRDKitが良いでしょう。どちらもデフォルトの設定で前編の問題設定の節で示した計算をやってみました。結果は以下のような図になります。OpenBabelとRDKitで計算されるSimilarityの値が一致する場合は赤線に乗ることになります。
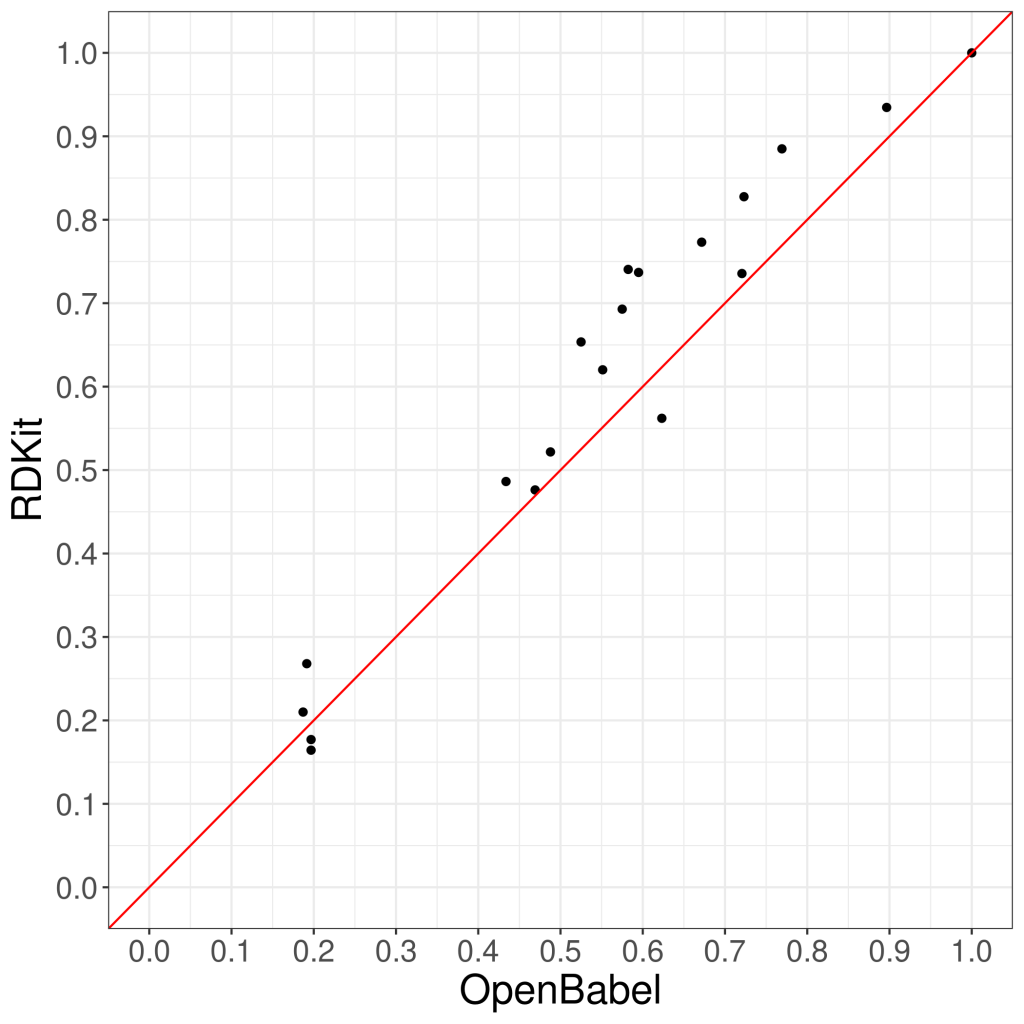
この図を見ると同じECFP4を利用したSimilarityであるにもかかわらず、結構バラツキがあるように見えます。想像されるバラツキの理由は、RDKitのECFP4のビット長とOpenBabelの方のビット長が異なることが考えられます。実際、Openbabelは4096ビットでRDKitの方は2048ビットになっています。しかしながらOpenBabelの方を2048ビットに変更して計算しても、多少数値が異なる化合物もあるという程度で、全く改善は見られなかったです。ちなみにビット数の変更は”GetFingerprint(&ref, fp_ref, 2048)”のように、フィンガープリントを計算するメソッドを呼び出す時に指定すれば良いです。
どうやら、OpenBabelとRDKitの間には計算方法に根本的な違いがあるかもしれません。想定以上にバラツキが大きい結果となりました。
最後に
今回はOpenBabelでSimilarity計算をするC++プログラムについて前後編に渡って紹介しました。結局Similarity計算にはOpenBabelとRDKitのどちらを利用すれば良いのでしょうか?それともこの程度のバラツキは許容範囲なのでしょうか?誰か知っていたら教えて頂けるとありがたいです。今後はOpenBabelだけでなく、RDKitについてもブログで紹介出来ればと考えています。
Category: Linux関連, OpenBabel, プログラミング関連