論文書いていたため、なかなかブログの更新していなかったのですが、久々にOpenBabelプログラミングネタのブログを投稿していこうと思います。今回はOpenBabelの分子力場を利用した計算をテーマにしたいと思います。
はじめに
分子力場と言えばケモインフォマティクスの超重要なテーマかと思います。分子の三次元構造を発生させるためには必ず分子力場が必要ですし、各種エネルギーを評価することができるので、色々な解析に応用できます。先ずは分子構造のエネルギー極小化するC++プログラムを紹介します。
プログラム
じつはOpenBabelにはエネルギー極小化のコマンドが標準でインストールされます。そのコマンド名は”obminimize”です。ここで紹介するC++プログラムはその”obminimize”コマンドのソースコードを可能な限りシンプルにしたようなものとなります。計算している内容は2次元のアラキドン酸の構造をエネルギー極小化して簡易的に3次元するというものです。
解説するプログラムとデータをまとめたZIPファイルをここからダウンロードしてください。C++コードの内容は以下の通りです。
1 #include <fstream>
2 #include <iostream>
3 #include <string>
4
5 #include <openbabel/mol.h>
6 #include <openbabel/obconversion.h>
7 #include <openbabel/forcefield.h>
8
9 using namespace std;
10 using namespace OpenBabel;
11
12 int main(int argc, char* argv[]) {
13 // 力場の設定
14 string ff = "MMFF94";
15 OBForceField* pFF = OBForceField::FindForceField(ff);
16 if (!pFF) {
17 cerr << "指定の力場が見つかりませんでした" << endl;
18 return -1;
19 }
20 pFF->SetLogFile(&cout);
21 pFF->SetLogLevel(OBFF_LOGLVL_LOW);
22
23 // ファイルオープン
24 ifstream ifs("Arachidonic_Acid.sdf");
25 if (ifs.fail()) {
26 cerr << "ファイルが見つかりませんでした" << endl;
27 return -1;
28 }
29
30 // 参照分子の読み込み
31 OBConversion conv;
32 conv.SetInAndOutFormats("SDF", "SDF");
33 OBMol mol;
34 conv.Read(&mol, &ifs);
35 ifs.close();
36
37 mol.AddHydrogens(); // 水素付加
38
39 // 分子をセットアップ(部分電荷などの計算)
40 if (!pFF->Setup(mol)) {
41 cerr << "分子の設定に失敗しました" << endl;
42 return -1;
43 }
44
45 // 最急降下法からの共役勾配法でエネルギー極小化
46 pFF->SteepestDescent(5000, 1e-1);
47 pFF->GetCoordinates(mol);
48 pFF->ConjugateGradients(5000);
49 pFF->GetCoordinates(mol);
50
51 // 分子を出力
52 ofstream ofs("Out_Arachidonic_Acid.sdf");
53 conv.Write(&mol, &ofs);
54 ofs.close();
55
56 return 0;
57 }このプログラムは以下のコマンドでコンパイルできます。なお、Anacondaを利用してOpenBabel3をインストールしてある必要があります。これについては以前のブログを参照してください。
g++ -std=c++11 Minimize.cpp -I${HOME}/anaconda3/envs/ob3/include/openbabel3 \
-L${HOME}/anaconda3/envs/ob3/lib \
-Wl,-rpath=${HOME}/anaconda3/envs/ob3/lib -l openbabelコードの解説
分子力場を利用するにあたり、includeすべきは7行目の””です。そして分子力場の利用開始時にOBForceFieldクラスのインスタンスを作成します。この処理は15行目で行っています。FindForceFieldというメソッドを利用することによりOpenBabelで分子力場パラメータセットを指定してOBForceFiledクラスのインスタンスが生成されます。14行目でff = "MMFF94"としているので、MMFF94という分子力場(低分子の計算では良く利用されます)を利用する例となります。
ちなみにOpenBabelでサポートされているパラメータセットは以下の表の通りです。
| 力場名 | 説明 |
|---|---|
| GAFF | General Amber Force Field (GAFF). |
| Ghemical | Ghemical force field. |
| MMFF94 | MMFF94 force field. |
| MMFF94s | MMFF94s force field. |
| UFF | Universal Force Field. |
20、21行目ではOpenBabelから出力されるログの設定をしています。ここではログの出力先を標準出力にして、ログの出力量をLOWに設定してます。OBFF_LOGLVL_NONEを設定するとログが出力されなくなります。続いて37行目では分子に水素原子を付加しています。40行目では分子をセットアップをしています。この処理で分子中の各原子の分子タイプや部分電荷をアサインしています。
このブログの主題のエネルギー極小化は46~49行目で処理を記述しています。OpenBabelでサポートしている最適化方法は最急降下法と共役勾配法です。それぞれの特徴ですが最適化方法は収束は遅いが頑健で、計算途中でエネルギーが発散するなど破綻することは稀です。一方、共役勾配法は収束は早いが時々計算が破綻することがあります。低分子の計算で破綻した経験は有りませんが、「タンパク質+大量の水分子」などの大きな系ではいきなり共役勾配法を適用すると破綻することがあります。多分、共役勾配法はエネルギー曲面を二次形式で近似することを前提にしていることが原因かなぁと思っています。つまり最急降下法である程度最適化して、エネルギー曲面が二次形式で近似しても大丈夫な感じにまでなったら共役勾配法を使うと良いでしょう。このプログラムでは最急降下法からの共役役勾配法で最適化しています。
最後に52~54行目で構造最適化した分子をファイルに出力しています。
計算結果
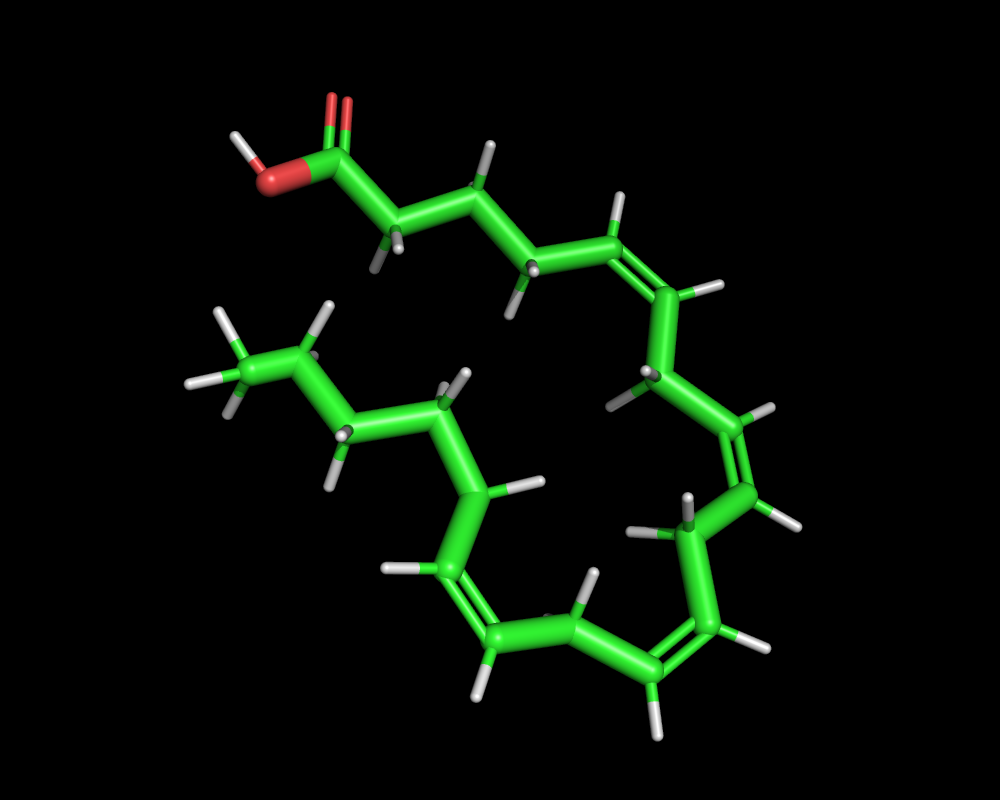
最後にエネルギー極小化したアラキドン酸の構造を以下に図示します。2次元構造をそのままエネルギー極小化しただけなのであまり変化を感じないですが、確かに3次元構造になっています。
Category: Linux関連, OpenBabel, プログラミング関連