前回に引き続き分子力場の話題の第二弾です。今回は最安定構造を探索してみようと思います。
はじめに
前回のブログでは単純にエネルギー極小化するプログラムを紹介しましたが、実用的な三次元分子構造を得るにはもっとエネルギーの低い構造を探索する必要があります。このような構造探索のことをコンホメーションサーチと言われます。OpenBabelにはコンホメーションサーチを実行する機能も付いているので、これを試してみようと思います。OpenBabelのC++のAPIドキュメントを見てみるとコンホメーションサーチの機能を提供するメソッドは次の表の通りです。
| メソッド名 | 説明 |
|---|---|
| SystematicRotorSearch | 回転可能な全ての結合を回転させる。組み合わせが爆発して実用的ではない。 |
| RandomRotorSearch | ランダムに選択された結合を回転させる。 |
| WeightedRotorSearch | エネルギーを低くするような結合の回転を優先的に計算する。 |
こうして見てみると”WeightedRotorSearch”が一番良いように見えます。実際”obabel”コマンドのオプションで”--gen3d best“と入力するとこの”WeightedRotorSearch”を利用して探索しています。ということで、このブログで試してみるコンホメーションサーチは”WeightedRotorSearch”としましょう。
プログラム解説
とりあえず、今回のブログのテーマのプログラムのコードを以下に示します。
1 #include <fstream>
2 #include <iostream>
3 #include <string>
4
5 #include <openbabel/mol.h>
6 #include <openbabel/obconversion.h>
7 #include <openbabel/forcefield.h>
8
9 using namespace std;
10 using namespace OpenBabel;
11
12 int main(int argc, char* argv[]) {
13 // 力場の設定
14 string ff = "MMFF94";
15 OBForceField* pFF = OBForceField::FindForceField(ff);
16 if (!pFF) {
17 cerr << "指定の力場が見つかりませんでした" << endl;
18 return -1;
19 }
20 pFF->SetLogFile(&cout);
21 pFF->SetLogLevel(OBFF_LOGLVL_LOW);
22
23 // ファイルオープン
24 ifstream ifs("Arachidonic_Acid.sdf");
25 if (ifs.fail()) {
26 cerr << "ファイルが見つかりませんでした" << endl;
27 return -1;
28 }
29
30 // 参照分子の読み込み
31 OBConversion conv;
32 conv.SetInAndOutFormats("SDF", "SDF");
33 OBMol mol;
34 conv.Read(&mol, &ifs);
35 ifs.close();
36
37 mol.AddHydrogens(); // 水素付加
38
39 // 分子をセットアップ(部分電荷などの計算)
40 if (!pFF->Setup(mol)) {
41 cerr << "分子の設定に失敗しました" << endl;
42 return -1;
43 }
44
45 // 最急降下法からの共役勾配法でエネルギー極小化
46 pFF->SteepestDescent(5000, 1e-1);
47 pFF->GetCoordinates(mol);
48 pFF->ConjugateGradients(5000);
49 pFF->GetCoordinates(mol);
50
51 pFF->WeightedRotorSearch(250, 10);
52 pFF->GetCoordinates(mol);
53
54 // 分子を出力
55 ofstream ofs("Out_Arachidonic_Acid.sdf");
56 conv.Write(&mol, &ofs);
57 ofs.close();
58
59 return 0;
60 }このプログラムは前回とほとんど同じです。51, 52行目にコンホメーションサーチする”WeightedRotorSearch”メソッドを利用する記述が挿入されただけです。
実に簡単ですね。実行して試したい場合は、前回のブログのプログラムを前述の通り修正してみてください。ちなみに引数の250と10はOpenBabelのソースコード(gen3d.cpp)の174行目の記述を参考にしています。
172 switch(speed) {
173 case 1:
174 pFF->WeightedRotorSearch(250, 10); // maybe based on # of rotatable bonds?
175 break;
176 case 2:
177 pFF->FastRotorSearch(true); // permute central rotors
178 break;
179 case 3:
180 default:
181 pFF->FastRotorSearch(false); // only one permutation
182 }計算結果
アラキドン酸のコンホメーションサーチを実行してみた結果の図を以下に示します。
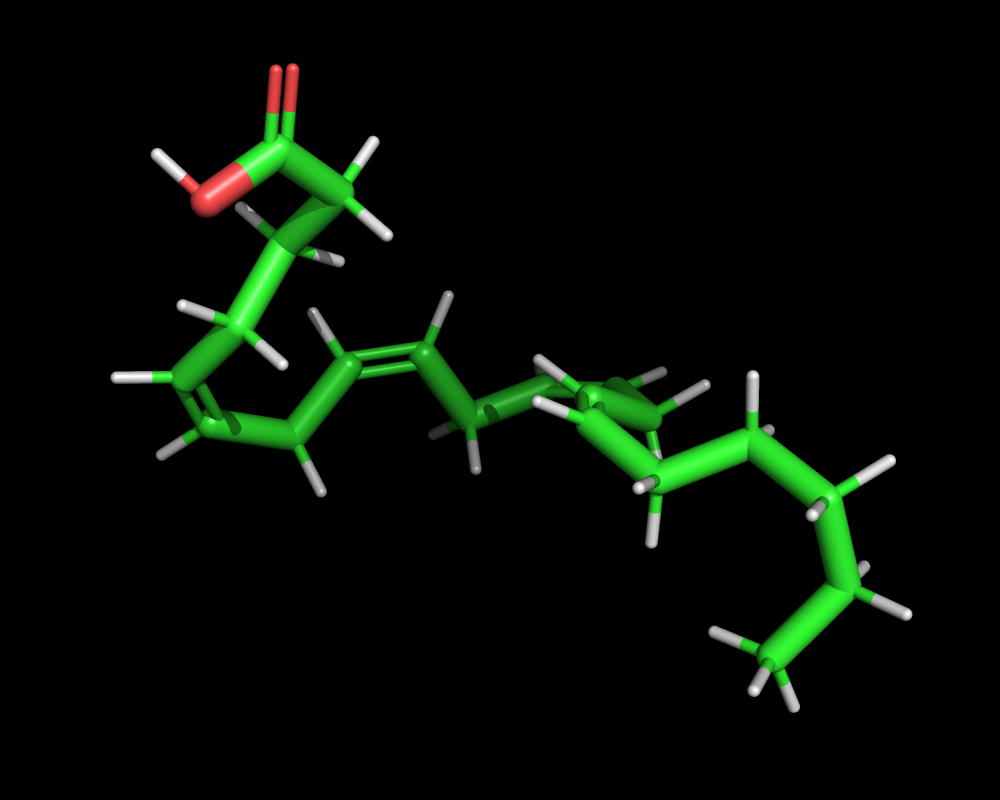
前回計算した結果と比べて中々良い感じの構造になったのではないかと思います。
飽和環を含む場合
上記のプログラムの結果は、OpenBabelの”--gen3d“オプションで3次元構造を立ち上げた場合と大体同じ結果になるのではないかと思います。で、このプログラムで飽和六員環を含む計算をすると少し残念な構造が出力される場合があります。例えばピペリジン環を含んでいるドネペジル(sdfile: Donepezil.sdf)を上記プログラムで三次元化すると、下図のように飽和六員環がねじれ舟型の構造になりました。
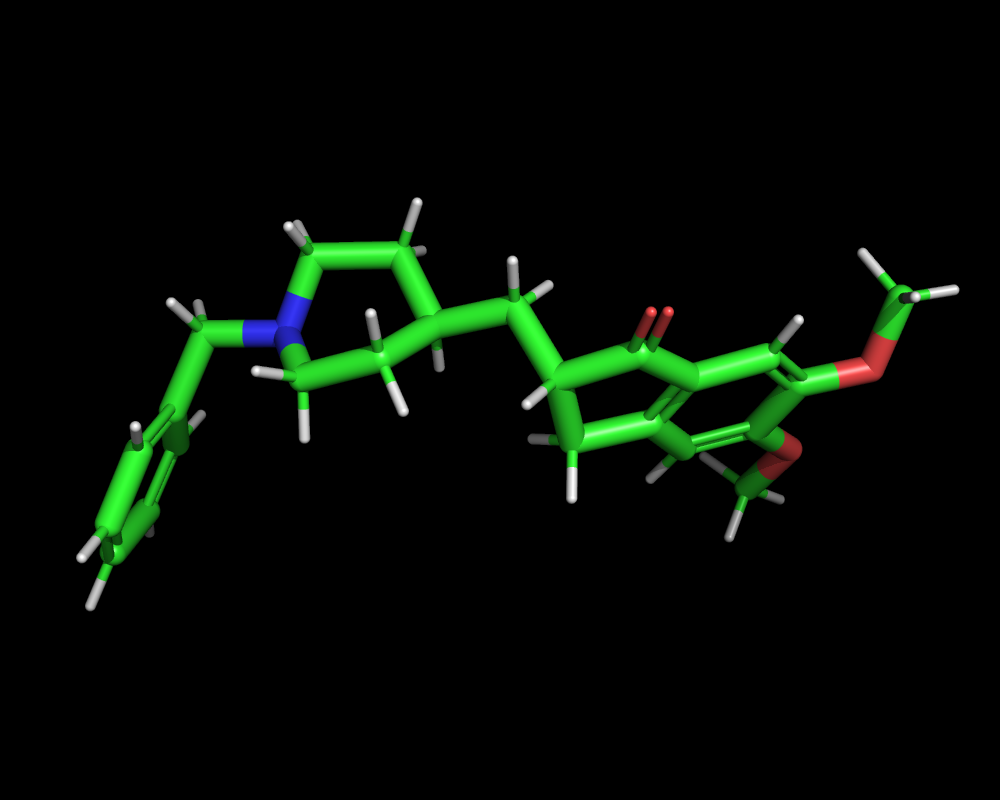
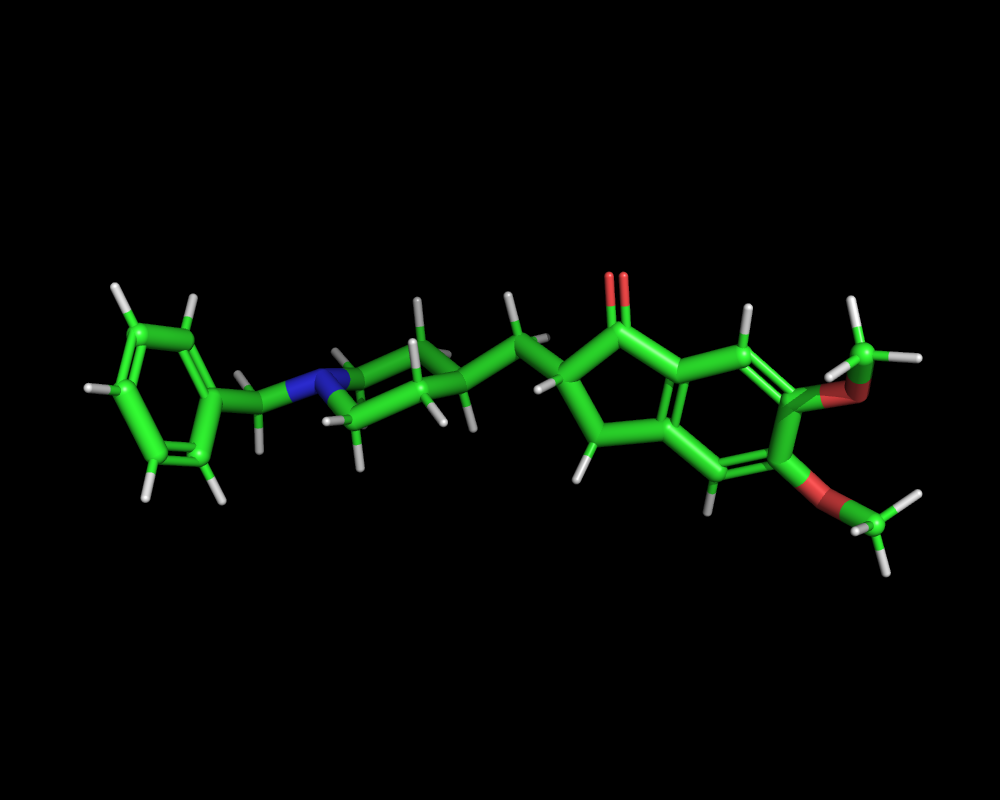
飽和六員環はいす型になる場合がほとんどのはずです。これは”WeightedRotorSearch”では環のコンホメーションまでは探索しない事が原因かと思われます。しかしながら、OpenBabelのAPIドキュメントを自分でビルドして読んでみると、オプション1つ入れるだけで環のコンホメーションも探索することができるようになっているみたいです。それは”WeightedRotorSearch”メソッドの第三引数に”true”を入れるだけです。ということでプログラムの51行目を以下のように微修正してみましょう。
51 pFF->WeightedRotorSearch(250, 10, true);このプログラムでドネペジルを三次元化すると下図のようないい感じのピペリジン構造になります。
Category: Linux関連, OpenBabel, プログラミング関連