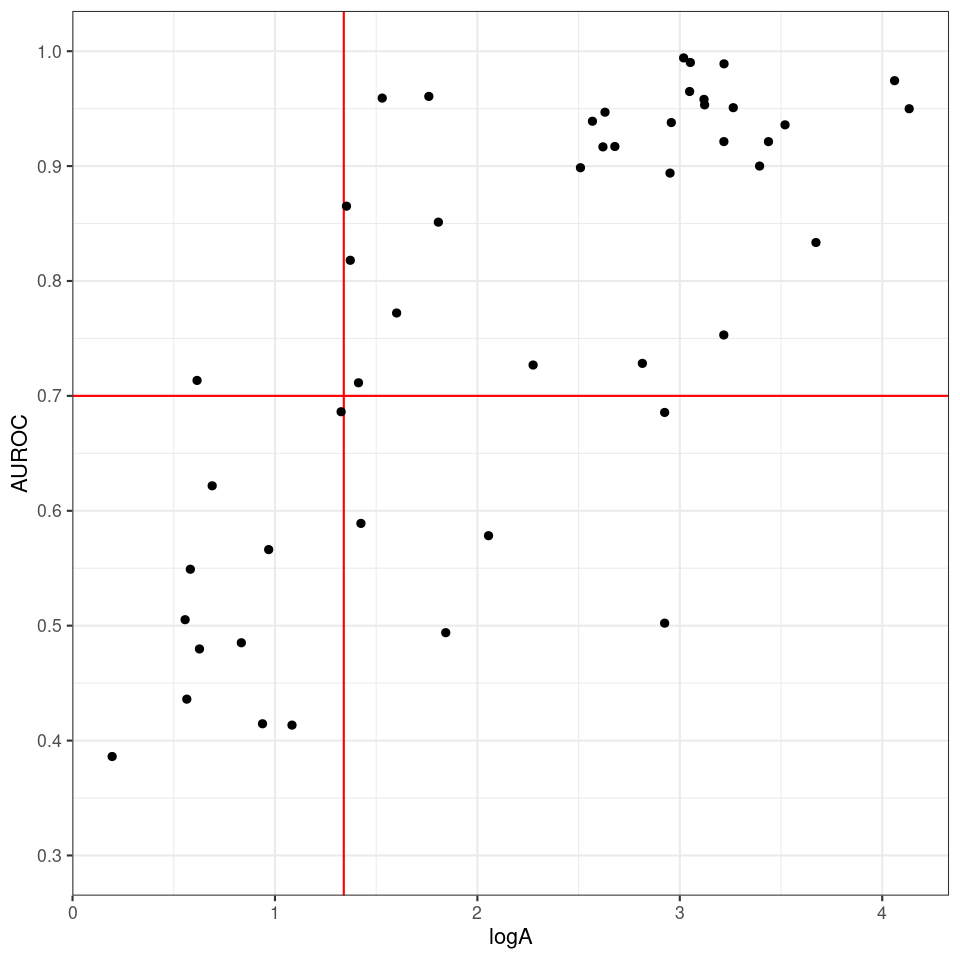
2021年8月に発表させていただいたCGBVS法の論文では、応用として学習データのないターゲットの予測可能性について検証しています。この論文ではデータのない標的タンパクに対する化合物スクリーニングの予測精度(AUROC)を推定する方法として、Applicability Index を定義しています。
今回は実際に我々の保有する予測モデル(Standardモデル)で、GPCR、KinaseについてApplicability Index を算出してみましたのでご紹介したいと思います。なお、論文で用いた予測モデルの学習データはChEMBL25をベースにしていますが、今回の予測モデルはChEMBL27のデータを利用しています。
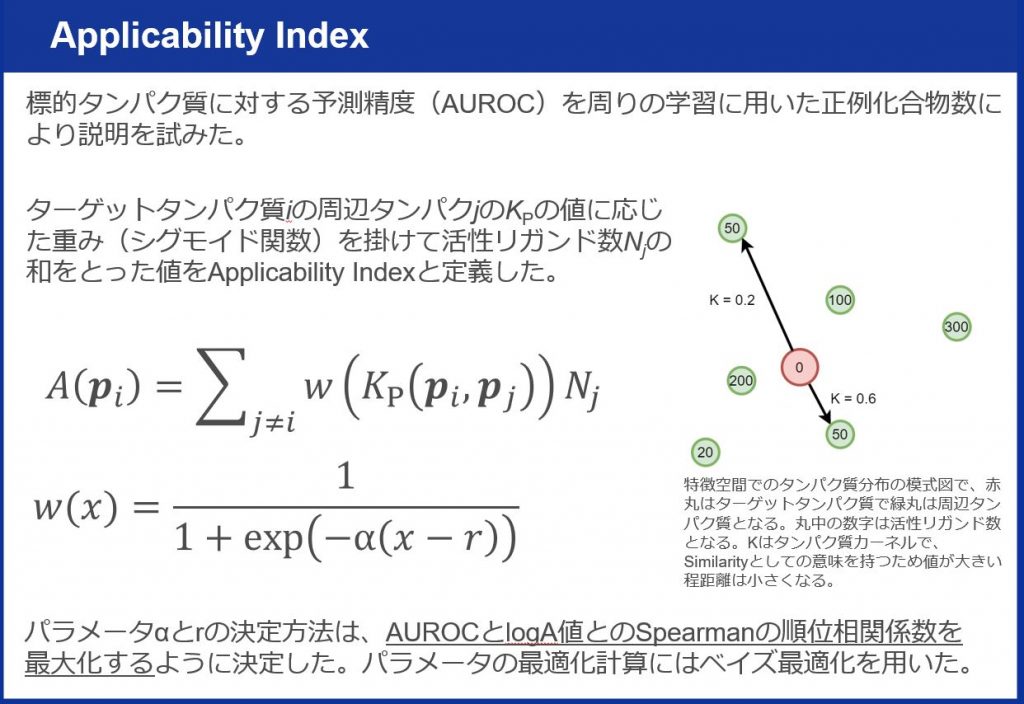
*Applicability Index については、以下のスライドを参照ください。
まず、学習データが全く無いオーファンとされるターゲットはGPCRで575ターゲット、Kinaseでは13ターゲット存在します。これらにそれぞれApplicability Index を算出したところ、AUROCが0.7以上となることが期待できる標的タンパク数がGPCRでは39ターゲット、Kinaseでは5ターゲット存在することが分かりました。
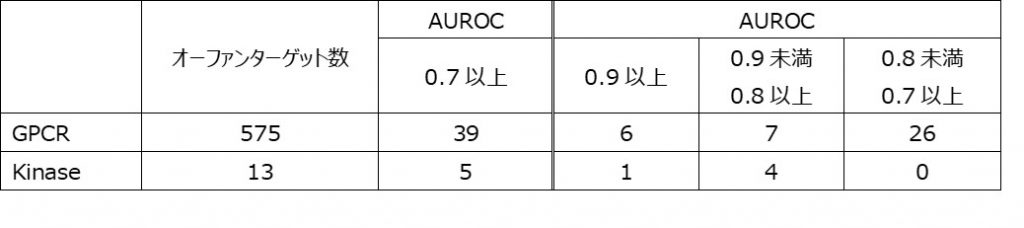
これらのターゲットについては、これまでCGBVSでは予測計算が不可能だろうとしておりましたが、GPCRでは39ターゲット、Kinaseでは7ターゲットについてはCGBVSにより、AUROCが0.7以上が期待できます。
—<補足>—
※各AUROCのThresholdは以下の値としています。

下図は計算した48個のGPCRとKinaseについてのlogA(Applicabilityの対数)とAUROCをプロットしたグラフです。
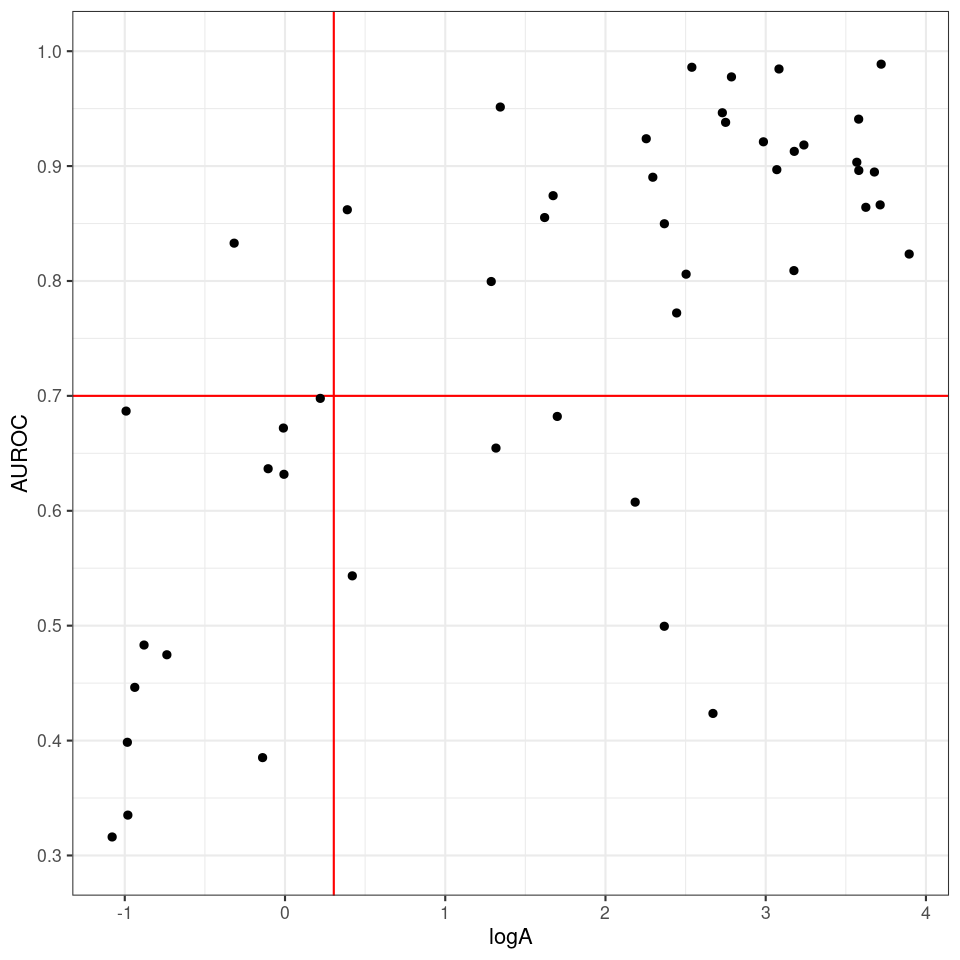
また今回は、学習データ(positiveデータ)が100個未満の標的ターゲットについてもApplicability Indexを算出してみました。それぞれGPCRでは45ターゲット、Kinaseでは190ターゲットとなります。これらは我々のHPで予測モデルのデータ数を公開していますが、これらのリストの△印のターゲットになります。
Applicability Index の結果は以下のようになりました。
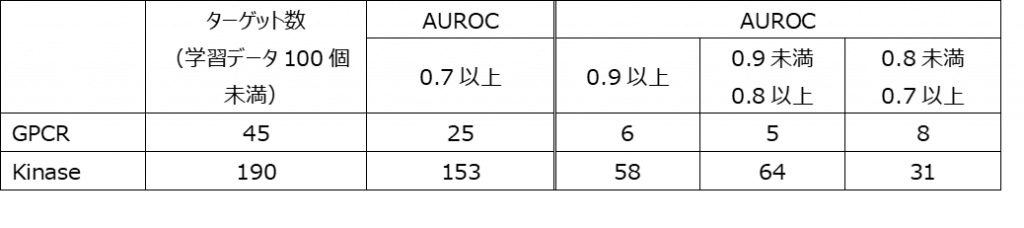
これまで、これらの学習データが100個未満のターゲットは、データ量が少ないことから、CGBVSでの予測計算は可能ではあるものの精度の面で不安がありました。ですが、今回のApplicability Index の結果をみますと、GPCRは45ターゲット中25ターゲット、Kinaseについては190ターゲット中153個が、AUROCが0.7以上となるような精度が期待できると言えそうです。
論文では、学習データのないオーファンターゲットの為にApplicability Index を定義しましたが、学習データが少ないターゲットに対しても、予測精度の信頼性を測る一つの指標として使えるものと思います。
今後、オーファンや予測モデルの主要ファミリーについては、それぞれApplicability Index を算出していく予定にしております。結果が出ましたらリスト化してHPで公開するなどお知らせできるようにしたいと思います。
追記(2021.11.09) GPCR、Kinase、Protease について、オーファンターゲットで計算可能なリストを公開しています。
Category: CGBVS/CzeekS, DRAGON/alvaDesc, Machine Learning