今回のブログもOpenBabelの分子力場の話題です。母核構造など、化合物分子の一部を固定してのコンホメーションサーチをやってみようと思います。
はじめに
前のブログではOpenBabelの分子力場のAPIを利用して低分子化合物のコンホメーションサーチを紹介しました。今回はそれを少し応用を加えて、母核構造を固定してコンホメーションサーチする方法を紹介します。この話のポイントは拘束条件の入れ方で、どのようにプログラムされるかを解説します。思ったよりも簡単にできますので、ぜひ最後まで読んでください。
母核固定してコンホメーションサーチする目的としては、化合物の側鎖部分をデザインして結晶構造の母核部分を重ねてみたときに、標的タンパク質のポケットに無理なく嵌るかどうかということが挙げられると思います。これを実現するにはプログラムなんぞ組まなくても、拘束条件付けてドッキングすれば良いだけなんだけど、拘束条件の入れ方が面倒だったり、そもそも拘束条件をサポートしていないドッキングツール(autodock vinaなど)があるので全く無意味でもないかと思ってます。
計算対象など
計算対象も前回のコンホメーションサーチから引き続きドネペジルとします。構造式は下図の通りです。母核構造は下図の青い部分のインダノンを指定することとします。
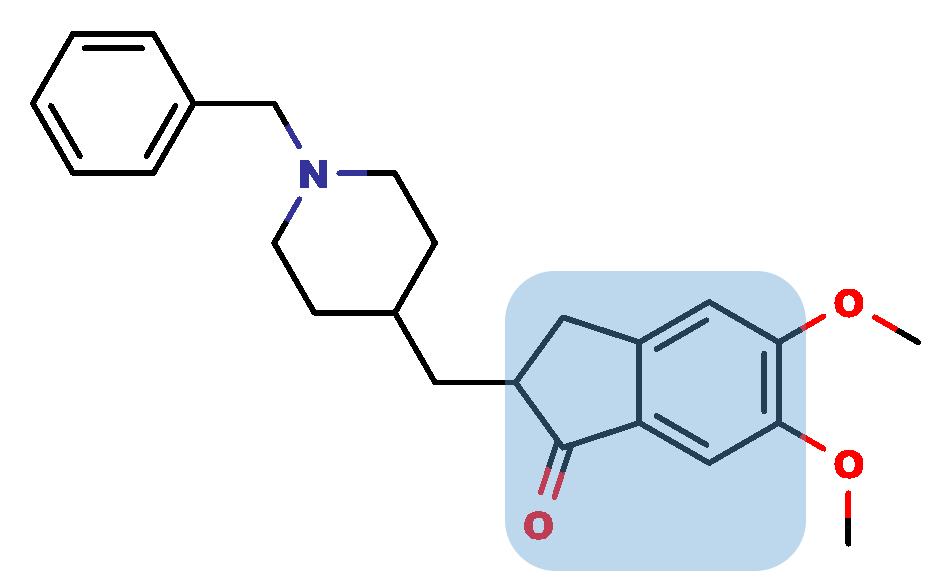
プログラムの仕様としては、側鎖部分のコンホメーションを10個発生させて、SDファイルとして出力するものとします。このとき、SDファイルのプロパティとして力場のエネルギー値を記述します。上記のような機能を持ったC++プログラムをなるべく簡潔に作成します。
プログラム解説
今回のプログラムとデータをまとめたZIPファイルをダウンロードしてください。少し長いですが、C++プログラムのコードを以下に示します。
1 #include <fstream>
2 #include <iostream>
3 #include <string>
4 #include <memory>
5
6 #include <openbabel/mol.h>
7 #include <openbabel/obconversion.h>
8 #include <openbabel/forcefield.h>
9 #include <openbabel/parsmart.h>
10 #include <openbabel/generic.h>
11
12 using namespace std;
13 using namespace OpenBabel;
14
15 int main(int argc, char* argv[]) {
16 // 力場の設定
17 string ff = "MMFF94";
18 OBForceField* pFF = OBForceField::FindForceField(ff);
19 if (!pFF) {
20 cerr << "指定の力場が見つかりませんでした" << endl;
21 return -1;
22 }
23 pFF->SetLogFile(&cout);
24 pFF->SetLogLevel(OBFF_LOGLVL_NONE);
25
26 // ファイルオープン
27 ifstream ifs("Donepezil.sdf");
28 if (ifs.fail()) {
29 cerr << "ファイルが見つかりませんでした" << endl;
30 return -1;
31 }
32
33 // 参照分子の読み込み
34 OBConversion conv;
35 conv.SetInAndOutFormats("SDF", "SDF");
36 OBMol mol;
37 conv.Read(&mol, &ifs);
38 ifs.close();
39
40 mol.DeleteHydrogens(); // 水素を外す
41
42 OBSmartsPattern smp;
43 smp.Init("O=C1CCc2c1cccc2");
44 smp.Match(mol);
45 vector<vector<int> > mlist = smp.GetUMapList();
46
47 // 拘束原子の設定
48 OBFFConstraints constraints;
49 for (int i = 0; i < mlist[0].size(); i++) {
50 constraints.AddAtomConstraint(mlist[0][i]);
51 }
52
53 // 分子をセットアップ(部分電荷などの計算)
54 if (!pFF->Setup(mol, constraints)) {
55 cerr << "分子の設定に失敗しました" << endl;
56 return -1;
57 }
58
59 pFF->RandomRotorSearch(10, 0);
60 pFF->GetConformers(mol);
61
62 mol.AddHydrogens();
63
64 char s[99];
65 ofstream ofs("out.sdf");
66 for (int i = 0; i < mol.NumConformers(); i++) {
67 mol.SetConformer(i);
68 if (!pFF->Setup(mol, constraints)) {
69 cerr << "分子の設定に失敗しました" << endl;
70 return -1;
71 }
72 pFF->ConjugateGradients(5000);
73 pFF->GetCoordinates(mol);
74 sprintf(s, "%f", pFF->Energy());
75 string Unit = pFF->GetUnit();
76 auto pd = new OBPairData;
77 pd->SetAttribute("Energy");
78 pd->SetValue(s);
79 mol.SetData(pd);
80 conv.Write(&mol, &ofs);
81 mol.DeleteData(pd);
82 }
83 ofs.close();
84
85 return 0;
86 }それでは解説したいと思います。1~39行目までは前回とほとんど同じです。ログの出力を無効化した程度の変化しかありません。そして40行目でわざわざ水素を削除しています。水素を削除しておかないとOpenBabelのコンホメーションサーチが正常に動作しないので、このような処理を入れています。ソースを見ても理由は良くわからなかったです。まあOpenBabelの仕様ということで諦めましょう(笑)。
次に42~45行目では母核構造で部分構造検索してマッチングしています。43行目のSMILES(SMARTS)で母核構造を具体的に指定しています。そして今回のテーマである拘束条件は47~51行目で設定しています。母核構造中の原子のみを拘束するために部分構造検索してマッチングしたわけです。54行目のSetupでOBFFConstraintsのインスタンスであるconstraintsを指定して、拘束条件を有効化します。次に59行目でランダムにコンホメーションを発生させています。このプログラムの例では10個発生させる設定です。この時点で発生させたコンホメーションはエネルギー的に無理のある構造も含まれますので、後ほどエネルギー極小化を行います。そして62行目で一度削除した水素を再び付加しています。
最後に66~82行目のループ内についての解説です。67行目でSetConformerメソッドを呼んで、molにi番目のコンホマーをセットしています。68行目で再び分子力場の拘束条件込みのセットアップをして、72行目で共役勾配法によるエネルギー極小化を実行しています。74~81行目でコンホマーのエネルギー値をSDファイルのプロパティとして設定して、80行目でファイルに出力しています。
このプログラムでは単純に原子の位置を固定する拘束条件ですが、設定できる条件には幾つか種類があります。以下の表に拘束条件に関わるメソッドをAPIドキュメントから抜粋してまとめました。
| メソッド名 | 説明 |
|---|---|
| SetFactor | 拘束力の強さを指定します。 |
| AddIgnore | エネルギーへの寄与を無視します。 |
| AddAtomConstraint | 原子位置を固定します。 |
| AddAtomXConstraint | 原子のX座標を固定します。 |
| AddAtomYConstraint | 原子のY座標を固定します。 |
| AddAtomZConstraint | 原子のZ座標を固定します。 |
| AddDistanceConstraint | 2原子間距離を拘束します。 |
| AddAngleConstraint | 3原子間の角度を拘束します。 |
| AddTorsionConstraint | 4原子間のねじれ角を拘束します。 |
| DeleteConstraint | 拘束条件を削除します。 |
計算結果
今回のプログラムのコンパイルは以下のようなコマンドになります。詳しくは以前のブログを参照してください。
g++ -std=c++11 PCS.cpp -I${HOME}/anaconda3/envs/ob3/include/openbabel3 \
-L${HOME}/anaconda3/envs/ob3/lib \
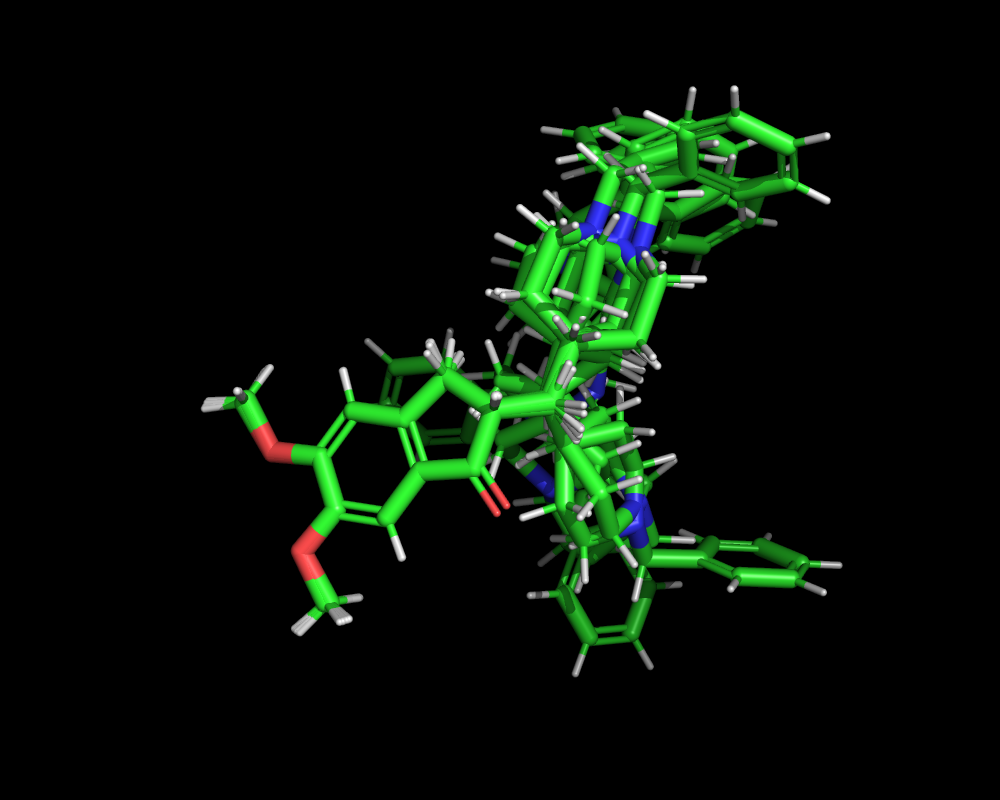
-Wl,-rpath=${HOME}/anaconda3/envs/ob3/lib -l openbabel$ ./a.outとしてプログラムを実行するとout.sdfというファイルが作成されていると思います。このSDファイルに母核構造を固定した10個の分子構造が入っているはずです。ちなみに乱数の系列が毎回異なるので、出力される構造も実行する度に異なります。私が計算した例を下図に示します。なかなか良い感じではないでしょうか?
Category: OpenBabel, プログラミング関連