今回の話題はタンパク質のポケット内にあるリガンドについてエネルギー極小化する方法についてです。特にタンパク質の構造を固定する場合についてどうすればよいか紹介したいと思います。あと、タンパク質とリガンドの力場エネルギー計算についても触れます。
はじめに
前回までのブログで自分で用意した化合物の母核部分をタンパク質ポケット内のリガンド分子の母核部分を重ね合わせるという計算を紹介しました。このような重ね合わせ計算をした後は相互作用エネルギーなどを計算してみたくなります。とりあえず化合物をタンパク質ポケット内に配置しただけだと、アミノ酸側鎖と化合物が衝突したりしてエネルギー的に高い状態にあることが普通です。このブログではタンパク質側を固定して化合物構造をエネルギー極小化してから、相互作用エネルギーを計算したいと思います。
タンパク質側を固定して化合物側をフリーにする設定は、以前のブログで紹介した母核部分を固定する方法を応用すれば基本的には良いです。しかしながら既定の設定では、固定部分のタンパク質は構成原子数が多いため、勾配の計算が必要ないのにもかかわらず計算されてしまいます。そして必要以上に多く計算時間がかかります。このことを避けるため、OpenBabelにおいてエネルギーを計算するグループを設定することができます。今回紹介するプログラムではエネルギーを計算するグループを設定する方法を用いています。
計算対象など
このブログで計算するのは前回から引き続きアセチルコリンエステラーゼとドネペジルです。このドネペジルについては結晶構造のものではなく、前回のブログで母核の重ね合わせ計算された構造を利用します。以下にエネルギー極小化計算を進めるタンパク質とリガンドの図を示します。
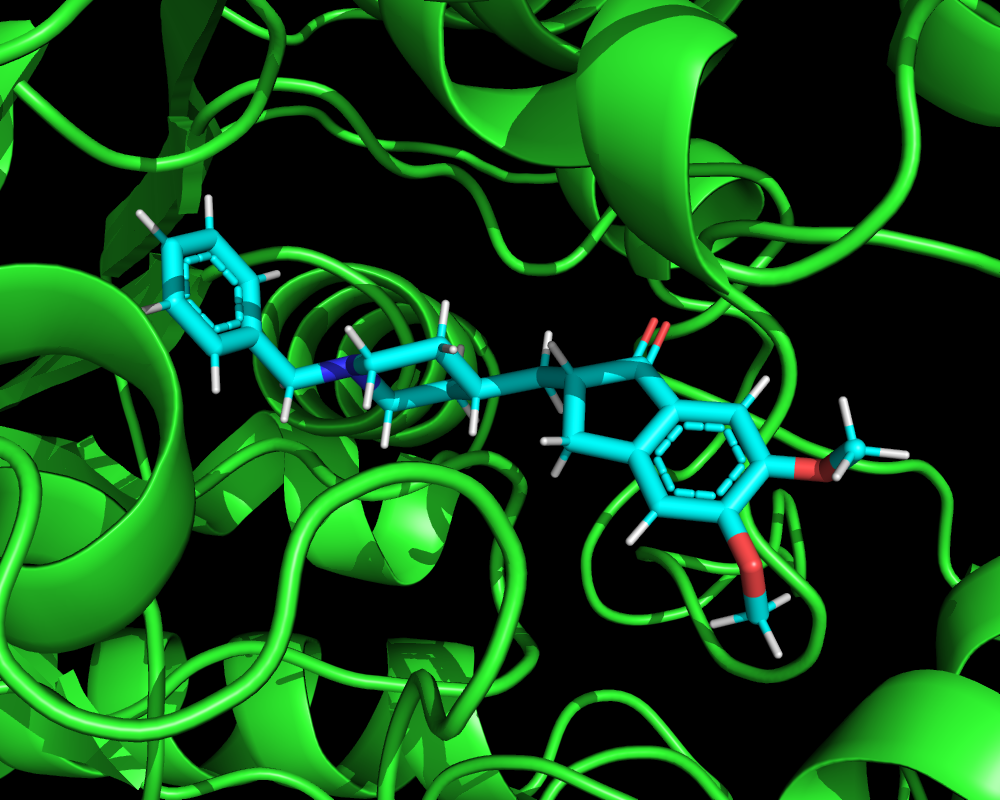
タンパク質側を固定してエネルギー極小化した後に、力場の相互作用エネルギーを評価してみましょう。
プログラム解説
今回のプログラムとデータをまとめたZIPファイルは以下のリンクからダウンロードしてください。
ZIPファイルを解凍すると3つのファイルが展開されます。Protein-Ligand.cppはC++のプログラムで、protein.pdbはタンパク質のPDBファイルです。そしてligand.sdfはリガンドのSDファイルです。プログラムのコンパイル方法は以下の通りです。
$ g++ -std=c++11 Protein-Ligand.cpp \
-I${HOME}/anaconda3/envs/ob3/include/openbabel3 \
-L${HOME}/anaconda3/envs/ob3/lib \
-Wl,-rpath=${HOME}/anaconda3/envs/ob3/lib -l openbabela.outというファイルが出来ていれば成功です。プログラムの実行は以下のコマンドです。
$ ./a.outそれではプログラムを見ていきましょう。C++のコードは以下の通りです。
1 #include <fstream>
2 #include <iostream>
3 #include <string>
4
5 #include <openbabel/mol.h>
6 #include <openbabel/obconversion.h>
7 #include <openbabel/forcefield.h>
8 #include <openbabel/parsmart.h>
9 #include <openbabel/generic.h>
10
11 using namespace std;
12 using namespace OpenBabel;
13
14 int main(int argc, char* argv[]) {
15 // 力場の設定
16 string ff = "MMFF94";
17 OBForceField* pFF = OBForceField::FindForceField(ff);
18 if (!pFF) {
19 cerr << "指定の力場が見つかりませんでした" << endl;
20 return -1;
21 }
22 pFF->SetLogFile(&cout);
23 pFF->SetLogLevel(OBFF_LOGLVL_LOW);
24
25 // ファイルオープン
26 ifstream ifs1("protein.pdb");
27 if (ifs1.fail()) {
28 cerr << "ファイルが見つかりませんでした" << endl;
29 return -1;
30 }
31
32 ifstream ifs2("ligand.sdf");
33 if (ifs2.fail()) {
34 cerr << "ファイルが見つかりませんでした" << endl;
35 return -1;
36 }
37
38 // タンパク質分子の読み込み
39 OBConversion conv;
40 conv.SetInAndOutFormats("PDB", "PDB");
41 OBMol mol;
42 conv.Read(&mol, &ifs1);
43 ifs1.close();
44
45 // 低分子リガンドの読み込み
46 OBConversion conv2;
47 conv2.SetInAndOutFormats("SDF", "SDF");
48 OBMol lig;
49 conv2.Read(&lig, &ifs2);
50 ifs2.close();
51
52 // リガンドの構成原子に印を付ける
53 for (auto ii = lig.BeginAtoms(); ii != lig.EndAtoms(); ii++) {
54 OBGenericData* pd = new OBPairData;
55 pd->SetAttribute("Ligand");
56 (*ii)->SetData(pd);
57 }
58
59 mol += lig; // タンパク質とリガンドを合わせる
60
61 // タンパク質とリガンドのグループの設定
62 OBBitVec protein(mol.NumAtoms());
63 OBBitVec ligand(mol.NumAtoms());
64 for (auto ii = mol.BeginAtoms(); ii != mol.EndAtoms(); ii++) {
65 int ix = (*ii)->GetIdx();
66 if ((*ii)->HasData("Ligand")) {
67 ligand.SetBitOn(ix);
68 protein.SetBitOff(ix);
69 } else {
70 ligand.SetBitOff(ix);
71 protein.SetBitOn(ix);
72 }
73 }
74
75 // タンパク質を固定する設定
76 OBFFConstraints constraints;
77 for (auto ii = mol.BeginAtoms(); ii != mol.EndAtoms(); ii++) {
78 if (protein.BitIsSet((*ii)->GetIdx())) constraints.AddAtomConstraint((*ii)->GetIdx());
79 }
80
81 // エネルギー計算のグループの設定
82 pFF->AddIntraGroup(ligand);
83 pFF->AddInterGroup(ligand);
84 pFF->AddInterGroups(ligand, protein);
85
86 // セットアップ
87 if (!pFF->Setup(mol, constraints)) {
88 cerr << "ERROR: could not setup force field." << endl;
89 }
90
91 // リガンド構造のエネルギー極小化
92 pFF->SteepestDescent(1000);
93 pFF->ConjugateGradients(1000);
94 pFF->GetCoordinates(mol);
95
96 // 相互作用エネルギーの計算
97 pFF->ClearGroups();
98 pFF->AddInterGroups(ligand, protein);
99 pFF->SetupCalculations();
100 cout << endl;
101 cout << "Interaction Energy = " << pFF->Energy() << " " << pFF->GetUnit() << endl;
102
103 // 結果の出力
104 ofstream ofs("out.pdb");
105 conv.Write(&mol, &ofs);
106 ofs.close();
107
108 return 0;
109 }先ず、15~23行目の力場の設定やログの設定は以前のブログで紹介した通りです。そして25~50行目まででタンパク質分子とリガンド分子を読み込んでいます。ここまでは特に新しい話題はありません。問題はこの後からです。52~57行目は原子にリガンドであるという印を付けています。具体的にはOBPairDataというクラスのインスタンスを各原子に登録しています。55行目でそのインスタンスに”Ligand”という属性をSetAttributeというメソッドでセットして、56行目でSetDataというメソッドを使って各原子にインスタンスを登録します。ちなみにOBPairDataクラスにはSetValueというメソッドもあって、任意の値を文字列として自由に登録できます。今回は”Ligand”という属性に対して登録すべき値は無いのでプログラムには書いていませんが、OpenBabelを使って分子に対して色々と細かい操作をするときに良く使います。RDKitにも似たようなクラスやメソッドがあるので、機会があれば紹介したいと思います。
59行目でタンパク質とリガンドをマージしています。OpenBabelでは分子のマージは+=を使います。61~73行目はタンパク質部分が1になるビット列とリガンド部分が1になるビット列を作成しています。ビット列はOBBitVecクラスを使って作成します。SetBitOnでビットを1にして、SetBitOffでビットを0にします。そして75~79行目でタンパク質を固定する拘束条件を設定しています。81~84行目でエネルギーを計算するグループを設定しています。
82行目は分子内の力場(結合している部分)エネルギーで、83行目は分子内のvdw相互作用と静電相互作用の計算する設定です。84行目は分子間、この場合はタンパク質とリガンド分子の設定です。そして設定の最後に87行目で力場のセットアップをしています。
91~94行目でエネルギー極小化計算をやっています。最終的に計算したいのはタンパク質とリガンドの相互作用エネルギーなので、分子内エネルギーの値は差し引きたいので、改めて97~99行目で分子間のエネルギーのみ計算するように、再設定を行っています。そして101行目で結果を標準出力しています。エネルギー極小化した構造も104~106行目でインスタンスconvを使うことによってPDB形式で出力しています。
計算結果
プログラムを動かすと色々と出力されて、最後は以下のような出力となります。
S E T T I N G U P C A L C U L A T I O N S
SETTING UP BOND CALCULATIONS...
SETTING UP ANGLE & STRETCH-BEND CALCULATIONS...
SETTING UP TORSION CALCULATIONS...
SETTING UP OOP CALCULATIONS...
SETTING UP VAN DER WAALS CALCULATIONS...
SETTING UP ELECTROSTATIC CALCULATIONS...
Interaction Energy = -19.6935 kcal/mol相互作用エネルギーは-19.6935 kcal/molとなりました。とりあえず負の値になって良かったです。あと、エネルギー極小化したリガンド構造は以下のような図になります。水色が計算前のリガンド構造で、オレンジ色が極小化後の構造です。こうして見ると母核部分はあまり動いていないことがわかります。
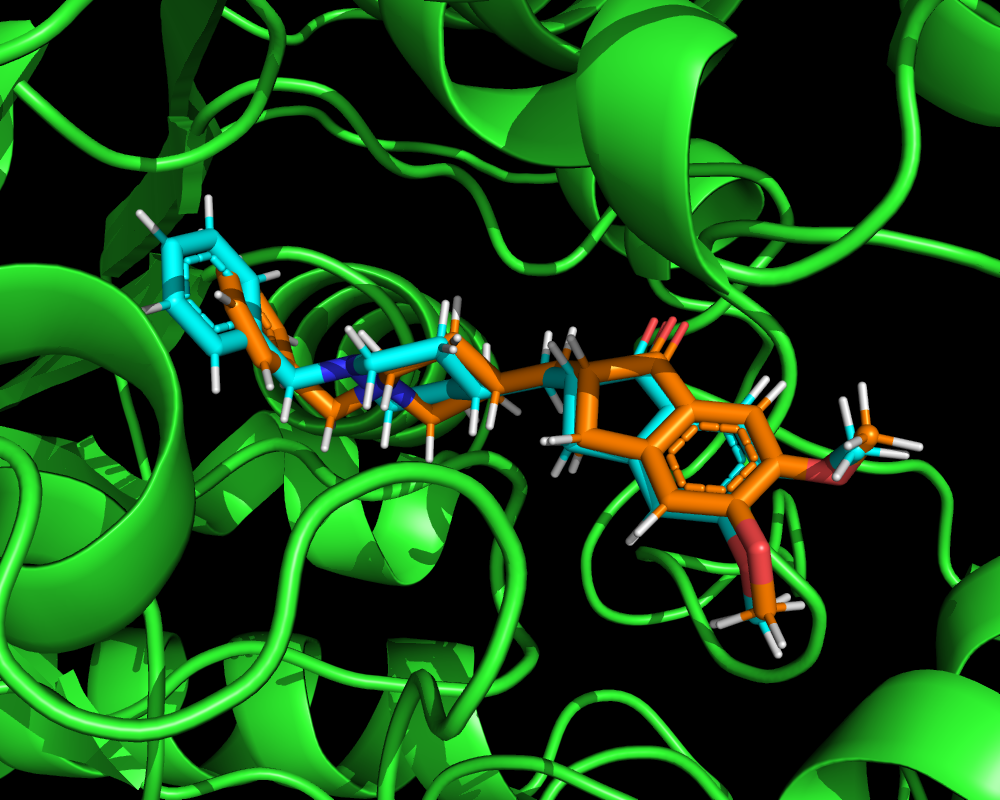
Category: OpenBabel, プログラミング関連