はじめに
このブログを始めた頃はPythonはあまり得意ではなく、専らC++でプログラムを書いてました。しかしながら、Pythonを使用せざるを得ないことが多く、私も大分Pythonに慣れてきました。私の最初のブログを見返してみると、「Pythonの勉強の成果みたいなこともブログにするかも」という記述がありました。今回は勉強の成果としてエクセルの化合物リストを作成するプログラムを紹介したいと思います。ちなみにSDファイルからエクセルファイルを作成するスクリプトはネット検索すれば幾つかヒットします。本格的に運用するのであれば、そちらの方を利用した方が良いかと思います。
動作環境の構築
Pythonのプログラムなので、anacondaかminicondaを利用して動作環境を構築します。前提としてAnacondaかMinicondaがインストールされているものとします。以下の内容のテキストファイルを作成します。
name: chemex
channels:
- rdkit
- defaults
dependencies:
- python=3.7
- openpyxl
- rdkitここでは”environment.yml”というファイル名で保存することとします。上記の内容をコピー&ペーストしてファイルを作成しましょう。そして以下のコマンドで仮想環境を作成しましょう。
$ conda env create -f environment.yml途中で何か聞かれたら”yes”と答えましょう。作成が終了したら以下のコマンドでPythonプログラムを実行する環境を立ち上げます。
$ conda activate chemexこれでプログラム実効する準備が整いました。
プログラムの実行
プログラムと入力データはこちらからダウンロードできます。
ダウンロードしたzipファイルを解凍するとPythonプログラムと化合物データファイルが現れます。化合物データはSDFとSMILESの2種類があります。上で説明した通り、condaコマンドで仮想環境を立ち上げてから以下のコマンドを実行してください。
$ python ChemEx.py sample1.sdf out.xlsxこのコマンドはSDFを入力する場合ですが、SMILESでも同じです。コマンドが正常動作した場合”out.xlsx”というエクセルファイルが作成されます。このファイルをエクセルで開くと下図のような化合物リストが見られると思います。
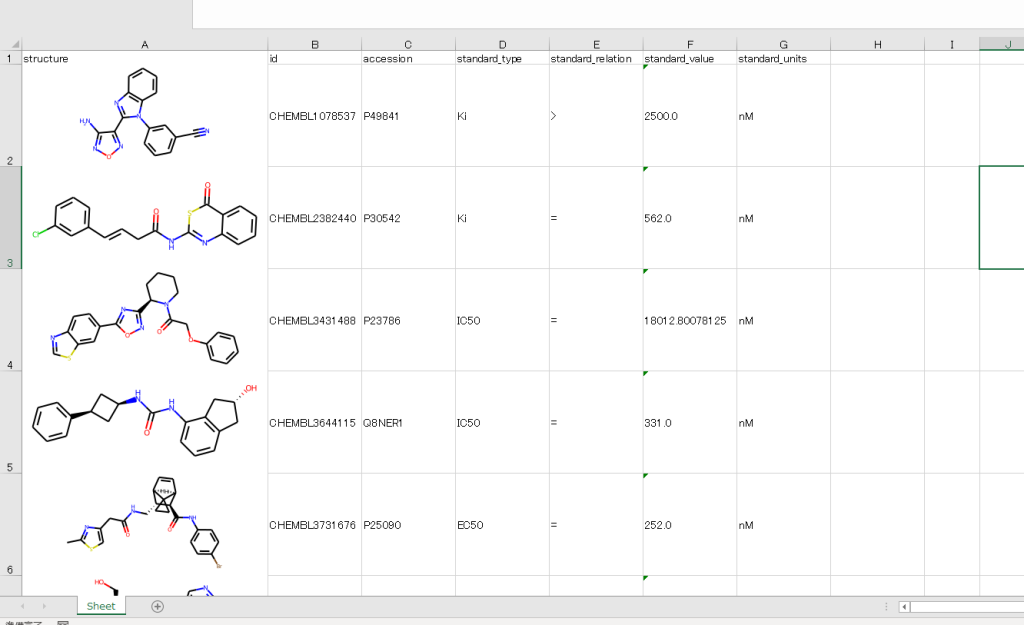
こんな感じでエクセルで化合物の構造式が確認できます。SDF中のプロパティを読み取ってリスト化しています。
プログラム解説
今回のプログラムは以下のとおりです。
1 #!/usr/bin/env python3
2 # coding: UTF-8
3 import sys
4 import os
5 import argparse
6 from rdkit import Chem
7 from rdkit.Chem import AllChem
8 from rdkit.Chem import Draw
9 import openpyxl
10 from openpyxl.styles.alignment import Alignment
11
12
13 # ファイルから分子を読み込みための処理をしてイテレータを返す。
14 def ChemSupplier(filename):
15 # 入力ファイルの存在チェック
16 if os.path.exists(filename) == False:
17 print("ファイルが存在しません")
18 quit()
19
20 # 拡張子を取得する
21 root, ext = os.path.splitext(filename)
22 ext = ext.lower()
23
24 # SDF or SMILESの読み込み設定
25 if ext == ".smi":
26 suppl = Chem.SmilesMolSupplier(filename, delimiter="\t", titleLine=True)
27 elif ext == ".sdf":
28 suppl = Chem.SDMolSupplier(filename)
29 else:
30 print("不明な拡張子です")
31 sys.exit()
32
33 return suppl
34
35
36 # 分子に付属するプロパティのリストを取得する。
37 def Propaties(suppl):
38 mol = next(suppl)
39 propnames = mol.GetPropNames()
40 suppl.reset() # supplを先頭に戻す
41
42 return propnames
43
44
45 if __name__ == "__main__":
46 p = argparse.ArgumentParser(description="sdfまたはsmiファイルからエクセル(xlsx)を作成します")
47 p.add_argument("input", help="input (sdf or smi)")
48 p.add_argument("output", help="output (xlsx)")
49 p.add_argument("-n", action="store_false", help="分子名のみ出力し、他のプロパティは出力しない")
50 args = p.parse_args()
51
52 suppl = ChemSupplier(args.input)
53 propnames = Propaties(suppl)
54
55 # excelファイル作成のためのワークブック生成
56 wb = openpyxl.Workbook()
57 ws = wb.worksheets[0]
58
59 # ヘッダーを記述します。
60 ws.cell(row=1, column=1).value = "structure" # 第1列は構造式とします。
61 ws.column_dimensions["A"].width = 40 # セルの幅を40にします。
62 ws.cell(row=1, column=2).value = "id" # 第2列は化合物IDとします。
63 # 第3列以降にSDファイルのプロパティを入れます。
64 for i, p in enumerate(propnames):
65 ws.cell(row=1, column=3 + i).value = p
66
67 for i, mol in enumerate(suppl):
68 # 構造式を画像ファイルを生成します。
69 mol = Chem.RemoveHs(mol)
70 AllChem.Compute2DCoords(mol)
71 img = Draw.MolToImage(mol, size=(320, 130))
72 # セルに画像を挿入します。
73 img = openpyxl.drawing.image.Image(img)
74 ws.row_dimensions[i + 2].height = 100
75 ws.add_image(img, ws.cell(row=i + 2, column=1).coordinate)
76
77 # 分子のIDを挿入します。
78 molname = mol.GetProp("_Name")
79 ws.cell(row=i + 2, column=2).value = molname
80 ws.cell(row=i + 2, column=2).alignment = Alignment(vertical="center")
81
82 # 分子のプロパティの値をセルに挿入します。
83 for j, p in enumerate(propnames):
84 val = mol.GetProp(p)
85 ws.cell(row=i + 2, column=3 + j).value = val
86 ws.cell(row=i + 2, column=3 + j).alignment = Alignment(vertical="center")
87
88 wb.save(args.output) # エクセルファイルに出力それでは簡単に解説したいと思います。最初の10行は利用するライブラリをインポートしています。そして14〜33行目はMDLSD形式かSMILES形式の化合物ファイルを引数として、化合物のイテレータを返す関数を定義しております。化合物のファイル形式はファイル名の拡張子から判断しています。37〜42行目はSDファイル中の先頭分子のプロパティリストを読み取っています。ここで読み取ったリストがエクセルファイルの列名となります。
このプログラムではエクセルファイルの操作に”openpyxl”を利用しています。56〜57行目はopenpyxlでワークブックとワークシートを新規作成しています。続く60〜65はエクセルシートの先頭行に列名を入れています。
67行目からイテレータから分子を1個づつ読み取って処理を開始します。69〜75行目は分子の2次元構造を生成して、画像データ(pillowの形式)を作成して、openpyxlの画像データ形式に変換して、エクセルシートの所定のセルに貼り付けています。ここのポイントは73行目のopenpyxlの画像データ形式に変換する部分です。Pythonで画像データやファイルを扱う場合は通常pillowライブラリを利用しますが、openpyxlではデータ変換しないとエラーになってしまいます。78〜80行目で分子のID(SDファイルの分子レコード毎の先頭行の文字列)をセルに入れています。83〜86行目は分子のすべてのプロパティの値をセルに入れています。最後に88行目でエクセルファイルに出力します。
まとめ
今回はC++ではなくPythonプログラムの紹介でした。さすがにC++でエクセルファイルを扱う便利なライブラリが見当たらない(無いことはないけど)ので、今回の内容はPythonだからこれだけシンプルに作成できるという事例だけ思います。私はPythonは未だ勉強中なので、今後も勉強がてらブログで成果を紹介できればと思います。
Category: RDKit, プログラミング関連