先日、環状ペプチドを低分子化するための”Peptide-to-Small Molecule”という手法が、国内の研究グループより発表されました。
Yoshida S, Uehara S, Kondo N, et al. Peptide-to-Small Molecule: A Pharmacophore-Guided Small Molecule Lead Generation Strategy from High-Affinity Macrocyclic Peptides.
J. Med. Chem.2022, 65(15):10655-10673.
https://doi.org/10.1021/acs.jmedchem.2c00919
この研究では、nicotinamide N-methyltransferase (NNMT)の活性を阻害する8残基からなる環状ペプチドの低分子化を実現しています。NNMTと環状ペプチドの結晶構造、並びに環状ペプチドのSAR情報等から、ファーマコフォアを導出している点に大きな特徴があると思います。このファーマコフォアをドッキングベースのバーチャルスクリーニングのフィルターとして用いて、Virtual Hit-Aを得ています。最終的には、cell-freeとcell-basedの両方で強い活性をもつ化合物(No. 14)のデザインに成功しています。環状ペプチドから導出したファーマコフォアが、低分子のデザインに大きく貢献しており、大変興味深い手法だと思います。
ACISSでは、ファーマコフォアを満たす化学構造を生成するフレームワーク「Deep Quartet」*1,2の開発を行っています。本稿では、まず上記の論文を参考に環状ペプチドのファーマコフォアの設定を行い、次に、Deep Quartetを用いて、このファーマコフォアを満たす化学構造の生成を行うことにします。
まずは、論文で定義されている4つのFeatureを参考に図1のファーマコフォアをLigandScoutを用いて設定しました。論文では水素結合ドナーのFeatureは、+電荷となっていますが、結合相手が主鎖のカルボニルのため、本稿では、水素結合ドナーとしました(ただし、このFeatureは、環状ペプチドのArg3に由来しているため、+電荷は阻害活性に重要なのかもしれません)。また、疎水Featureの位置が、論文とは若干異なっているかと思いますが、とりあえず、図1のファーマコフォアを使ってみたいと思います。結晶構造は、論文と同じPDB(ID:7WMC)を利用しています。
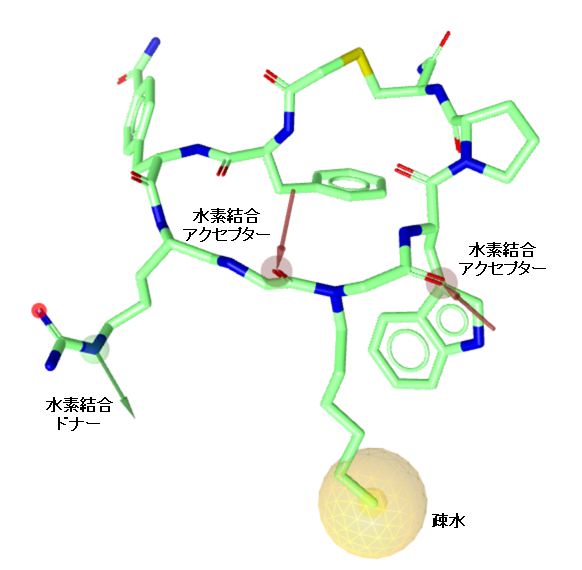
図1のファーマコフォアを使って、Deep Quartetを実行しました。学習のステップとファーマコフォアスコアの関係を図2に示します。
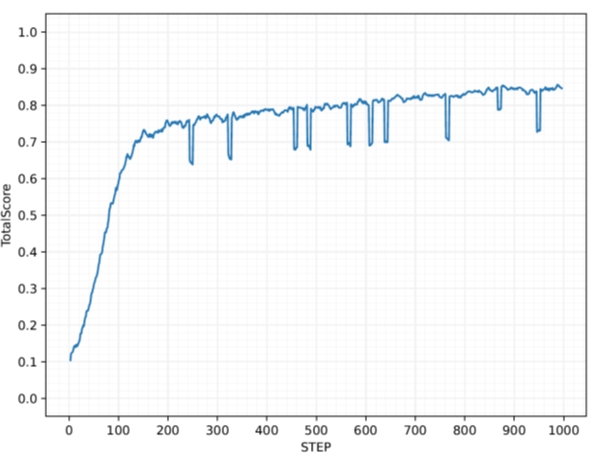
500 STEPくらいで、ファーマコフォアスコア(平均)が0.8を超えており、適切に学習が進んでいることが分かります。最終的にDeep Quartetで生成された化合物の1つ(化合物A)を図3に示します。
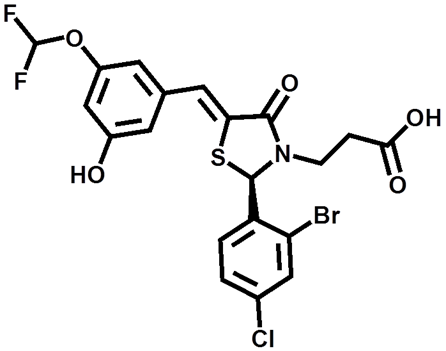
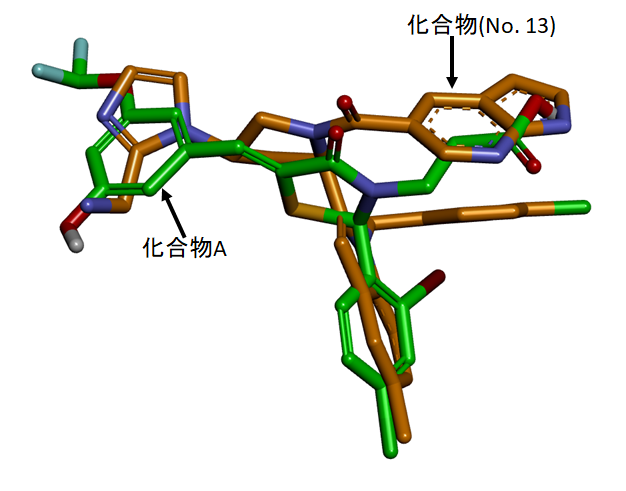
環状ペプチドと化合物Aの重ね合わせを図4に示します。
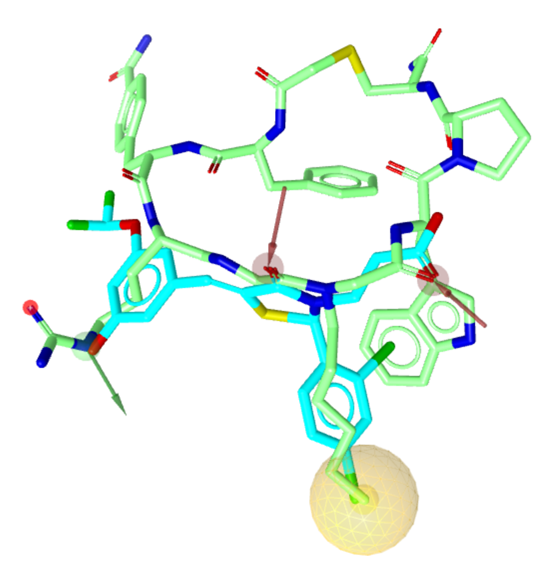
化合物Aのファーマコフォアスコアは、0.96(ファーマコフォアスコアの最大は1.0)であり、設定したファーマコフォアをほぼ完全に満たしています。Feature間の距離が少し長いため、構造生成には難しいファーマコフォアだと思いましたが、Deep Quartetによりファーマコフォアを満たす化学構造の生成が無事にできました。
論文中でデザインされた化合物(No. 13)とNNMTの結晶構造は、PDB(ID:7WMT)に公開されています。最後に、化合物Aと化合物(No. 13)の重ね合わせを図5に示します。論文を参考にファーマコフォアを設定しましたので、化合物Aと化合物(No. 13)の水素結合ドナー、アクセプター、疎水基の位置は、ほぼ一致しています。ただし、疎水Featureについては、もう少し調整が必要だと考えてます。
本稿では、論文で発表された環状ペプチドのファーマコフォア情報を用いて、Deep Quartetを実行してみました。ファーマコフォアを適切に設定できるケースにおいては、環状ペプチドの低分子化においても、Deep Quartetが有効に機能するかもしれません。
Category: AI創薬関連, Deep Quartet