今回から化合物の部分構造検索についての話題を取り扱います。これが出来るようになると、OpenBabelを利用したプログラミングが段々楽しくなってきます。先ずは入門的なプログラムを紹介しようと思います。
SMARTS
SMARTSとは化学構造を検索するためのクエリ記述言語です。これは構造式を一行で記述するSMILESを拡張したもので、元々はDaylight社が開発しました。現在では幾つか派生SMARTS(方言)が有るみたいです。Daylight社によるオリジナルのSMARTSの仕様を確認する場合は以下のリンクをクリックして下さい。
ここではSMARTS自体の解説まではしません。SMARTSなんて初めて聞いたという方は、恐らくDaylight社のページを見ても良く分からないかもしれません。そういう場合はChemAxon社のMarvinSketchという構造描画ツールを使ってみると良いでしょう。このツールが保存形式にSMARTSを選択することが出来ます。つまりMarvinSketchで検索したい構造と検索条件を描画して、SMARTS形式でファイル保存して、そのファイルをテキストエディタで内容を確認すれば良いのです。
単純な部分構造検索
簡単なサンプルプログラムを以下に示します。ファイル名は”SSS1.cpp“です。リンクをクリックしてダウンロードしてみて下さい。
1 #include <iostream>
2 #include <fstream>
3 #include <string>
4
5 #include <openbabel/mol.h>
6 #include <openbabel/obconversion.h>
7 #include <openbabel/parsmart.h> // SMARTSを利用する場合はこれをincludeする
8 #include <openbabel/mcdlutil.h>
9
10 using namespace std;
11 using namespace OpenBabel;
12
13 int main(int argc, char* argv[]) {
14 OBConversion conv;
15 conv.SetInAndOutFormats("SMI", "SDF");
16
17 OBMol mol;
18 conv.ReadString(&mol, "COc1cc2ncnc(c2cc1OCCCN1CCOCC1)Nc1ccc(c(c1)Cl)F");
19 generateDiagram(&mol);
20 mol.SetDimension(2);
21
22 OBSmartsPattern smp; // パターンマッチするためのクラスを宣言する
23 smp.Init("c1ccccc1"); // SMARTSパターンの翻訳
24 smp.Match(mol); // パターンマッチの実行
25 vector<vector<int> > mlist = smp.GetUMapList(); // 実行結果の取得
26
27 for (int i = 0; i < mlist.size(); i++) {
28 cout << "match" << i + 1 << ":";
29 for (int j = 0; j < mlist[i].size(); j++) {
30 cout << " " << mlist[i][j];
31 }
32 cout << endl;
33 }
34 }このプログラムでは分子標的薬の一つであるゲフィチニブを例にとり、ベンゼン環をクエリとして部分構造検索しています。18行目でゲフィチニブのSMILESから分子構築しています。そして一連の部分構造検索は22〜25行目で行っています。検索するためのクエリは23行目で設定していて、”c1ccccc1″はベンゼン環のSMARTSとなります。検索結果は整数のvectorのvectorで返されます。つまり2次元配列と言うことですね。もしもマッチす部分構造が無かった場合はサイズ0のvectorになります。また、24行目のMatchメソッドの返り値がfalseになります。プログラム自体は単純ですよね。問題はクエリであるSMARTSを如何に書くかが難しいのですが、これは慣れるしか無いですね。
上記プログラムを以下の様にコマンドでコンパイルして、実行します。
$ g++ -std=c++11 SSS1.cpp -I/home/username/app/include/openbabel-2.0 -L/home/username/app/lib -lopenbabel
$ ./a.out
match1: 3 12 11 10 5 4
match2: 24 29 28 27 26 25出力されるのは、match1とmatch2についての2種類の数列ですが、その意味はクエリにマッチしたゲフィチニブの原子インデックス番号のことです。クエリが6個の炭素原子からなるベンゼン環(通常水素原子はクエリには含めない)なので、数字が6個あります。この例題の場合、ゲフィチニブの原子インデックス番号は下図の様になっています。この番号は同一化合物であってもSDファイルやSMILESの書き方によって異なりますので注意が必要です
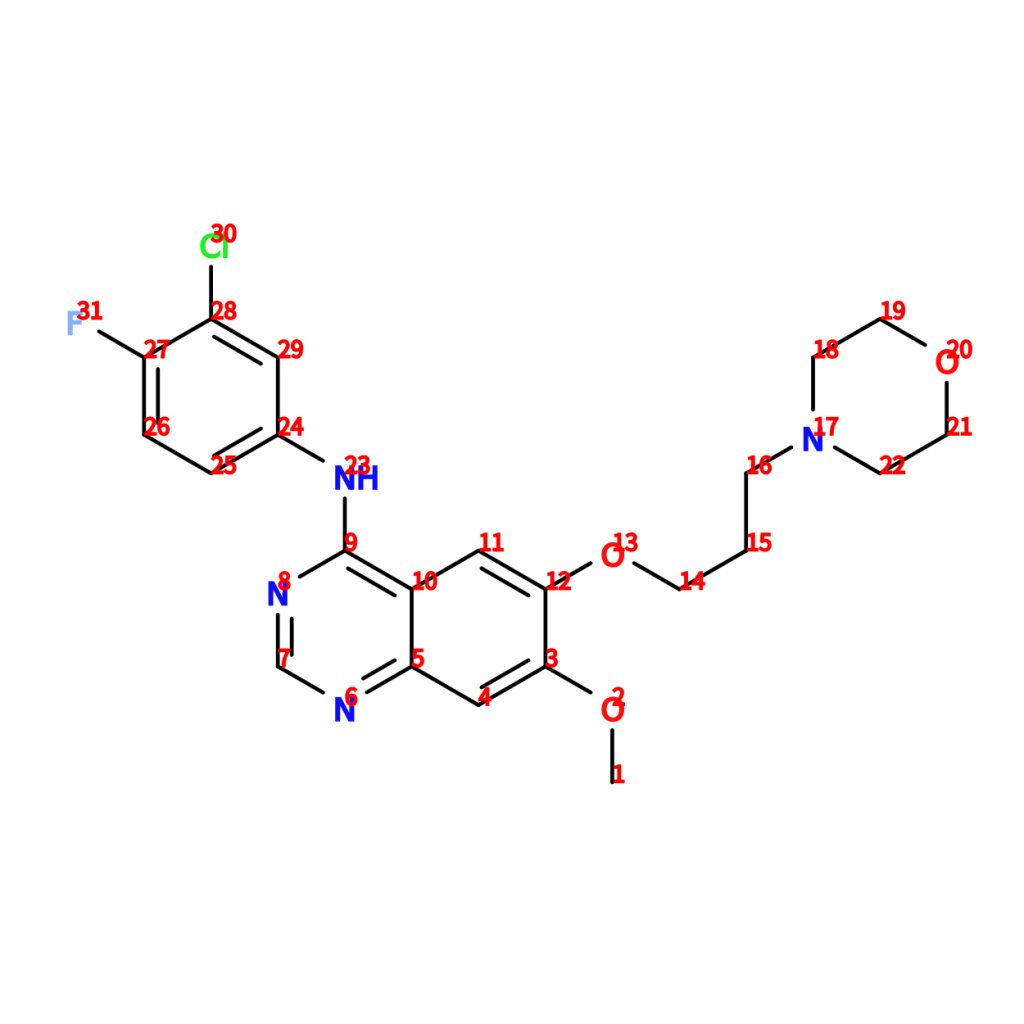
以上より、match1のインデックス番号は3、12、11、10、5、4なので、ゲフィチニブ中のキナゾリン環のベンゼン部分が対応します。そしてmatch2のインデックス番号は24、29、28、27、26、25なので、ゲフィチニブ中のフッ素と塩素が付いているベンゼン環部分が対応します。
ちょっとした応用
部分構造検索を利用した例として、ゲフィチニブのモルホリン環をチオモルホリン環に変換するプログラムを紹介します。ソースコードは以下の通りです。ファイル名は”SSS2.cpp“です。リンクをクリックしてダウンロードしてみて下さい。
1 #include <iostream>
2 #include <fstream>
3 #include <string>
4
5 #include <openbabel/mol.h>
6 #include <openbabel/obconversion.h>
7 #include <openbabel/parsmart.h>
8 #include <openbabel/mcdlutil.h>
9
10 using namespace std;
11 using namespace OpenBabel;
12
13 int main(int argc, char* argv[]) {
14 OBConversion conv;
15 conv.SetInAndOutFormats("SMI", "SDF");
16
17 OBMol mol;
18 conv.ReadString(&mol, "COc1cc2ncnc(c2cc1OCCCN1CCOCC1)Nc1ccc(c(c1)Cl)F");
19 generateDiagram(&mol);
20 mol.SetDimension(2);
21
22 OBSmartsPattern smp;
23 smp.Init("C1COCCN1");
24 smp.Match(mol);
25 vector<vector<int> > mlist = smp.GetUMapList();
26
27 mol.BeginModify();
28 OBAtom* a = mol.GetAtom(mlist[0][2]);
29 a->SetAtomicNum(16);
30 mol.EndModify();
31
32 conv.Write(&mol, &cout);
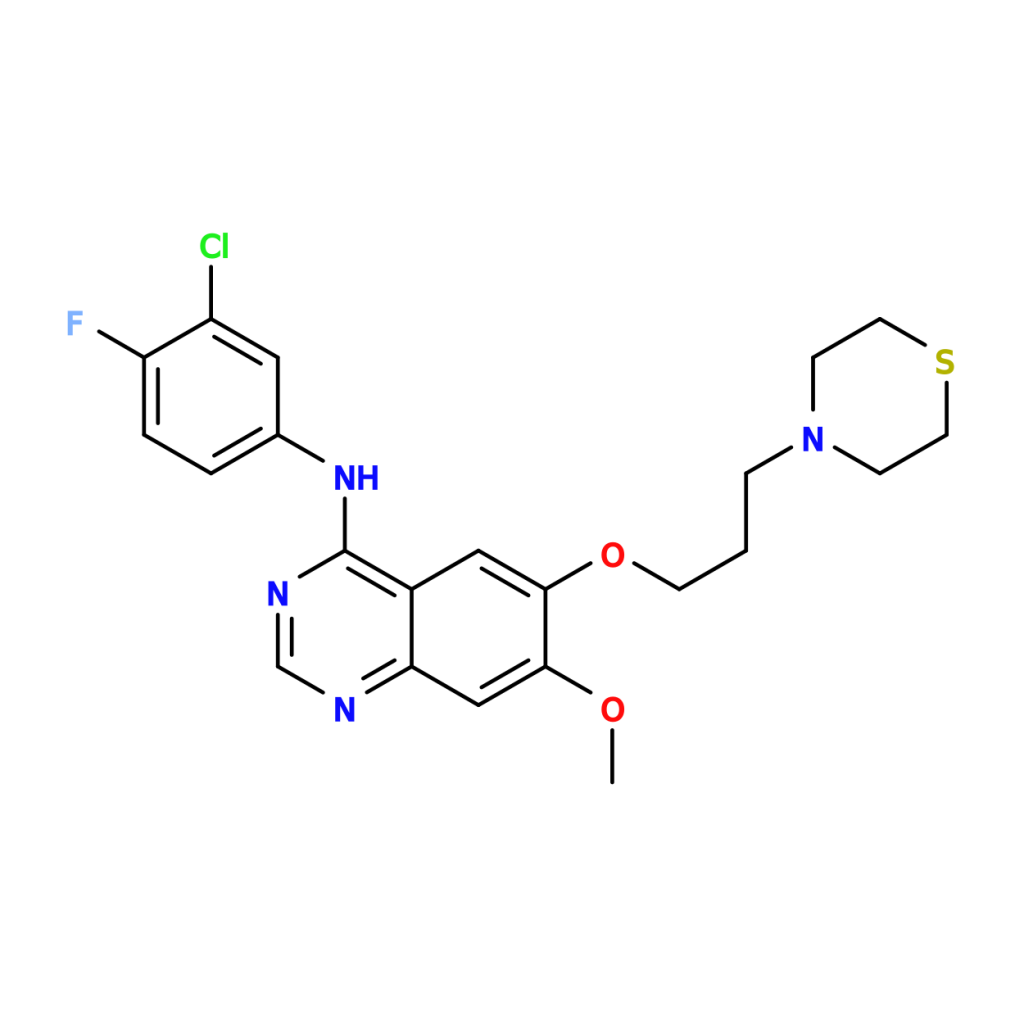
33 }最初のプログラムとはクエリが異なりモルホリン”C1COCCN1″になっています(23行目)。そしてマッチした結果を利用してモルホリン環の酸素原子を硫黄原子に置換している部分が27〜30行目です。OpenBabelで分子を操作するときは、最初にBeginModify()を実行してから分子操作を行って、最後にEndModify()を実行します。SQLのBEGIN〜COMMITみたいなものですね。28行目で変換対象の酸素原子へのポインタを取得しているのですが、このときのmlist[0][2]の”0″と”2″の意味ですが、先ず”0″はマッチした最初の部分構造という意味です。ゲフィチニブの場合はモルホリン環が1個だけなので0としていますが、複数ある場合はforループを利用するなどします。次に”2″はクエリである”C1COCCN1″の3番目の原子に対応しているという意味です。つまりmlist[0][2]にはモルホリン環中の酸素原子のインデックス番号が入っています。29行目の操作で当該原子の原子番号を16(硫黄)にセットして変換しています。そして最後に32行目でSD形式として分子を標準出力します。
このプログラムを以下のようにコンパイルと実行をします。出力はリダイレクトして”out.sdf”というSDファイルにしています。
$ g++ -std=c++11 SSS2.cpp -I/home/username/app/include/openbabel-2.0 -L/home/username/app/lib -lopenbabel
$ ./a.out > out.sdf出力結果を図にすると以下のようになります。
Category: OpenBabel, プログラミング関連