今回は、LigandScoutを用いたファーマコフォアの構築とバーチャルスクリーニングを行ってみます。
例として、DUD-EからPARP1の阻害剤とそのデコイを取得してバーチャルスクリーニングを実施します。DUD-Eは、ドッキングソフトウェアのベンチマークを行うためのデータベースであり、102の標的タンパク質に対して22,886の活性分子が登録されています。さらに1つの活性分子あたり50個のデコイ分子が登録されています。まずは、ここからactives_final.sdf.gzとdecoys_final.sdf.gzをダウンロードします。
Step 1: PARP1と阻害剤の複合体構造の取得
LigandScoutを起動し、PDB 4-letter codeである”3l3m”を入力して右側のボタンを押すとPDBからデータを取得することができます。
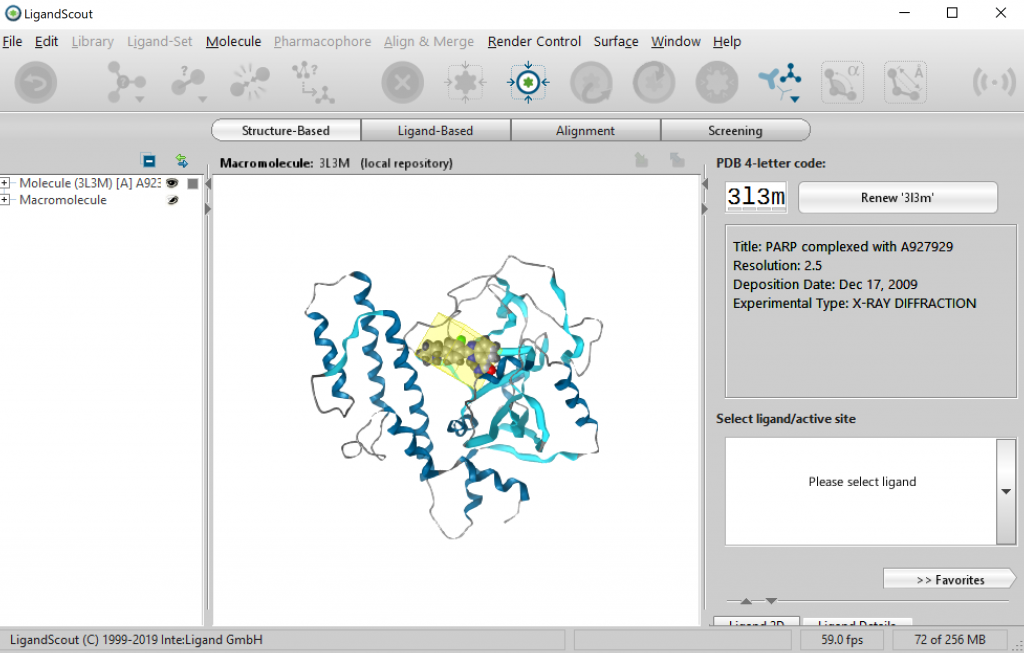
Step 2: ファーマコフォアの構築
阻害剤の周辺にある黄色の四角で囲まれている領域をクリックします。
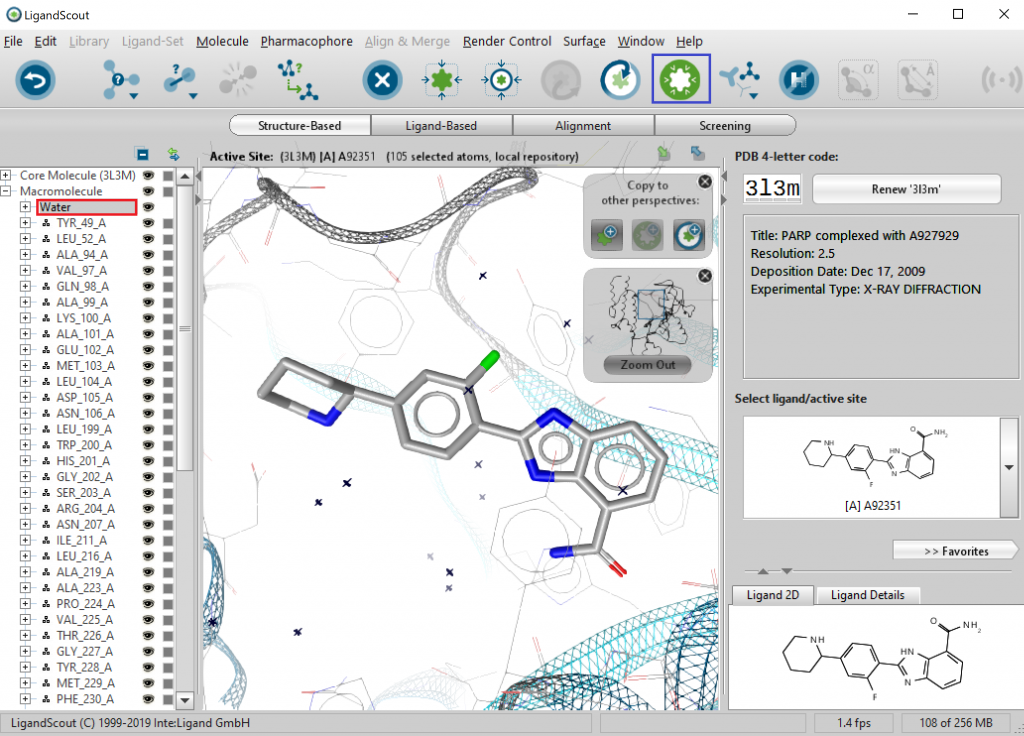
Water(赤四角)を選択しdeleteキーを押すことで水分子を削除します。その後、ファーマコフォアを構築するためにpharmacophoreアイコン(青四角)を押します。
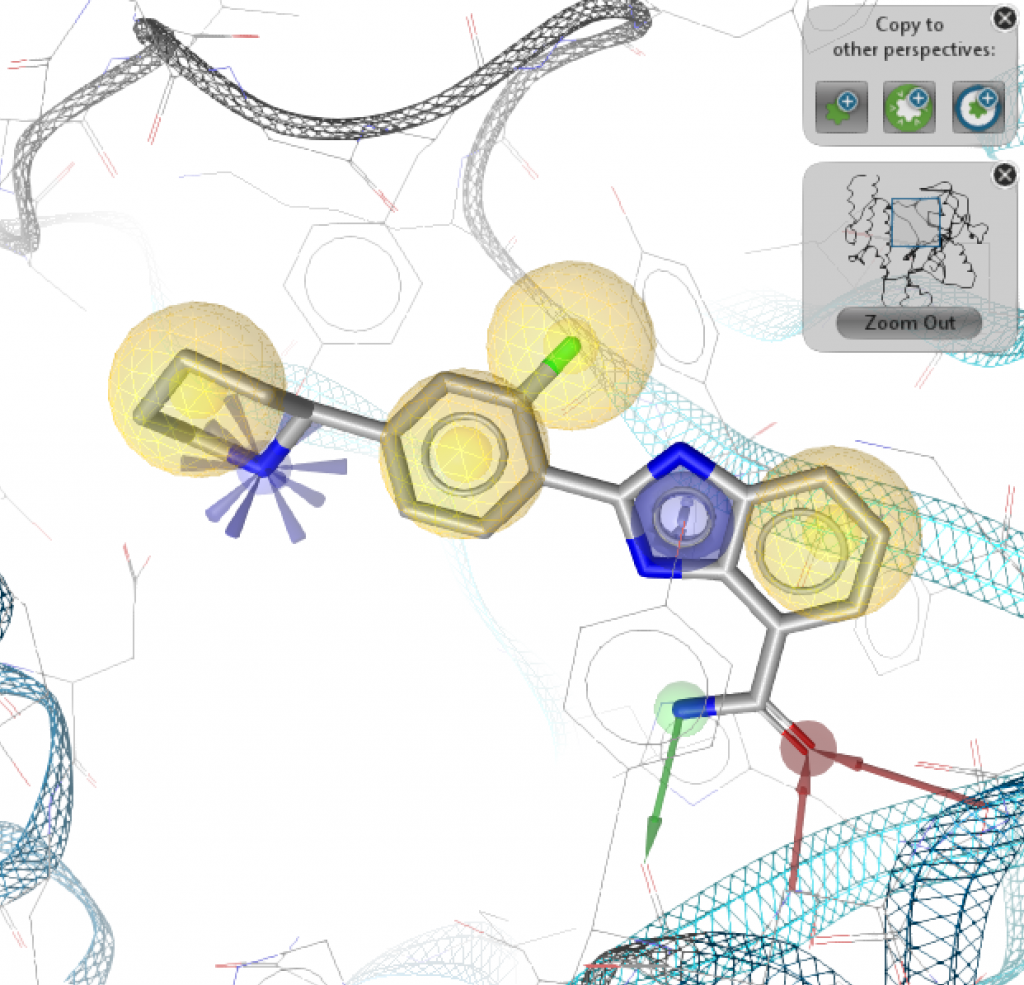
ファーマコフォアの構築ができました。Copy to other perspectivesの真ん中のボタンを押して、screening perspectiveを選択します。
Step 3: 活性分子とデコイ分子の配座生成
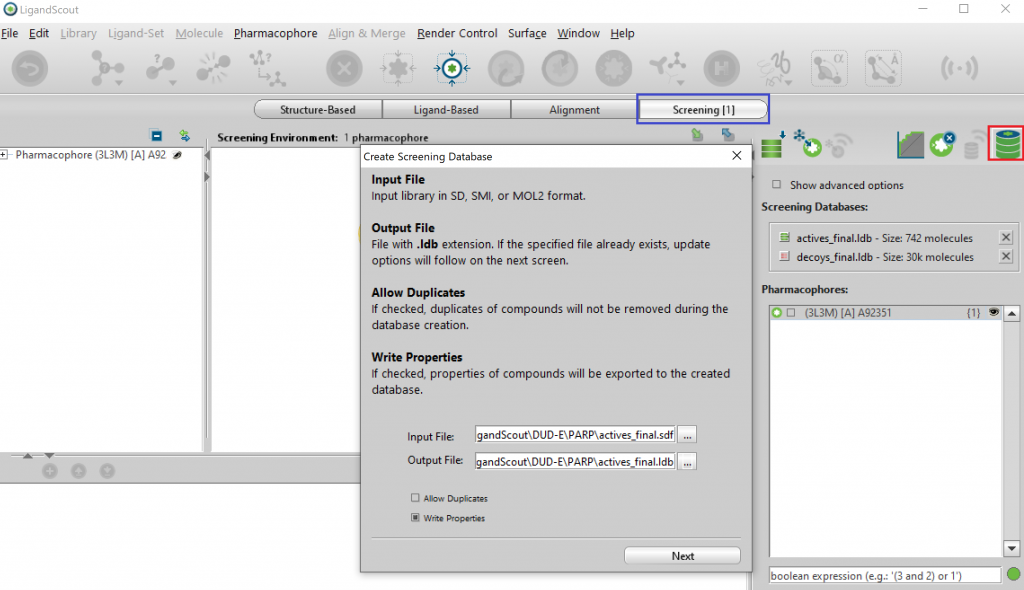
Screeningボタン(青四角)を押しScreening perspectiveに移動します。その後、右上のデータベースアイコン(赤四角)を押します。Create Screening Database windowが開きますので、Input FileをDUD-Eから取得したactives_final.sdfとし、Output Fileをactives_final.ldbとしてNextボタンを押します。本操作により配座生成を行うことができます。デコイ分子も同様の操作を行います。
Step 4: バーチャルスクリーニング
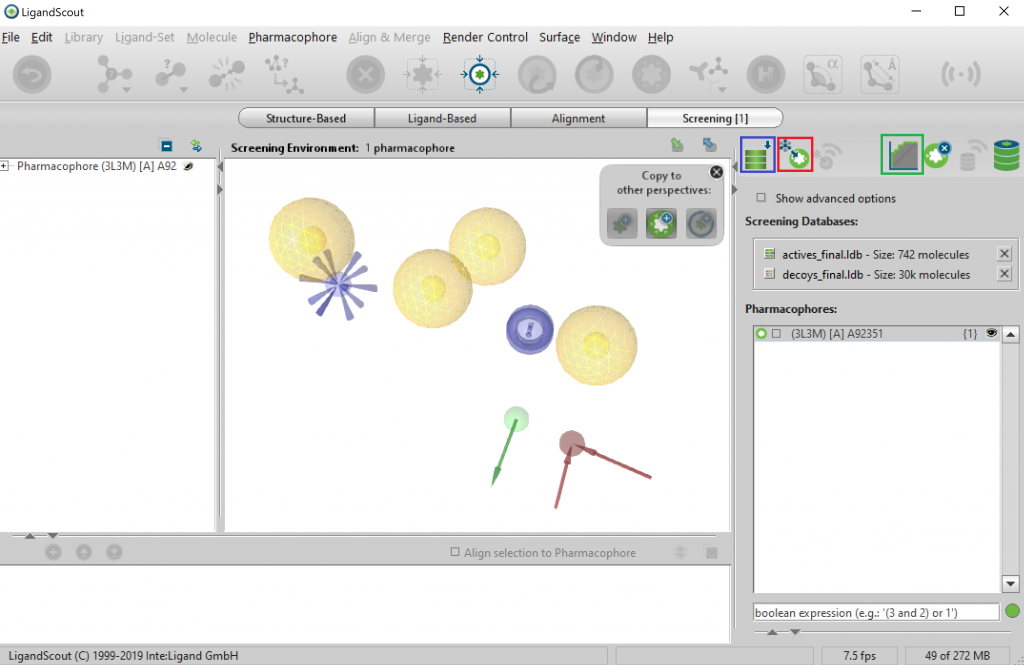
青四角内のアイコンを押して、Step 3で作成したactives_final.ldbとdecoys_final.ldbを入力します。actives_final.ldbの左側のマークを緑(活性分子)、そして、decoys.final.ldbの左側のマークを赤(デコイ分子)にします。
次に、赤四角内のアイコンを押すことでバーチャルスクリーニングがスタートします。スクリーニングが終わるまでしばらく待ちます。
スクリーニング終了後、緑四角内のアイコンを押すことでスクリーニング精度の評価をAUC、EFにより実施することができます。
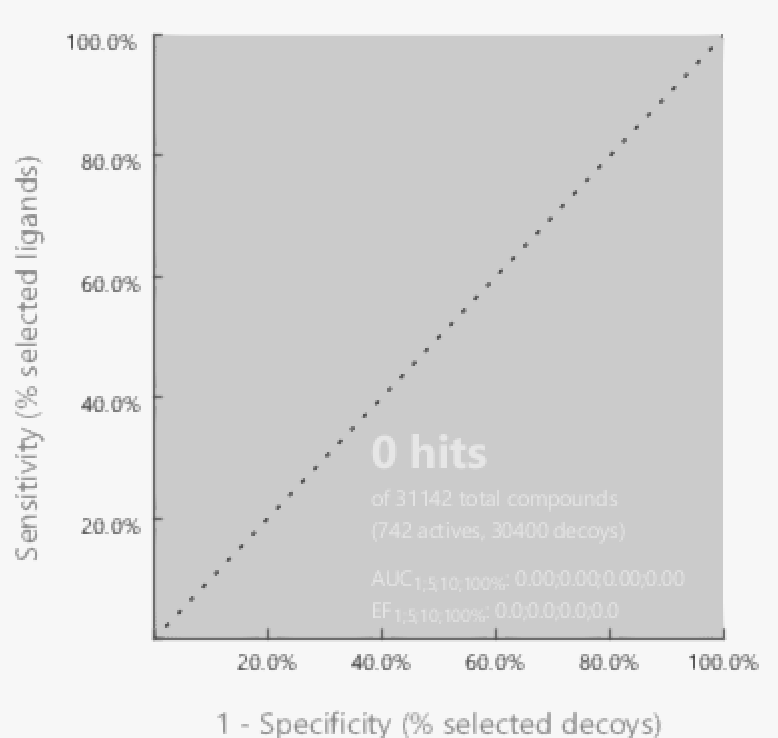
残念ながらヒット化合物はゼロでした。さすがにファーマコフォアを構成する8個のFeatureを全て満たす分子は無いようです。
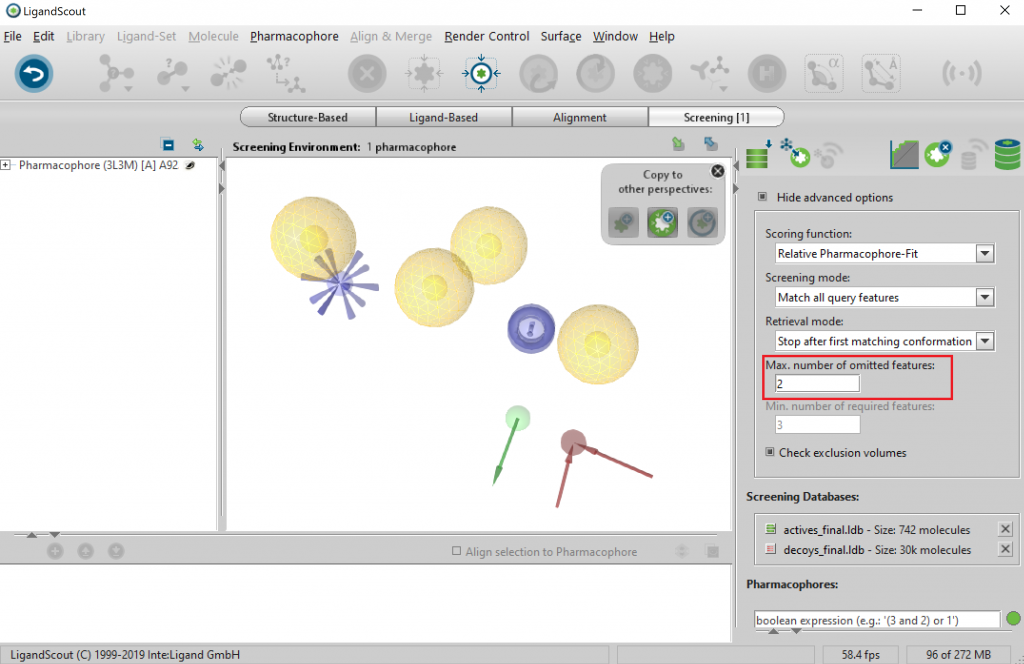
そこで、Hide advanced optionsを表示し、Max. number of omitted features (赤四角)を2とします。これで少なくとも6個のFeatureが満たされればヒットすることになります。
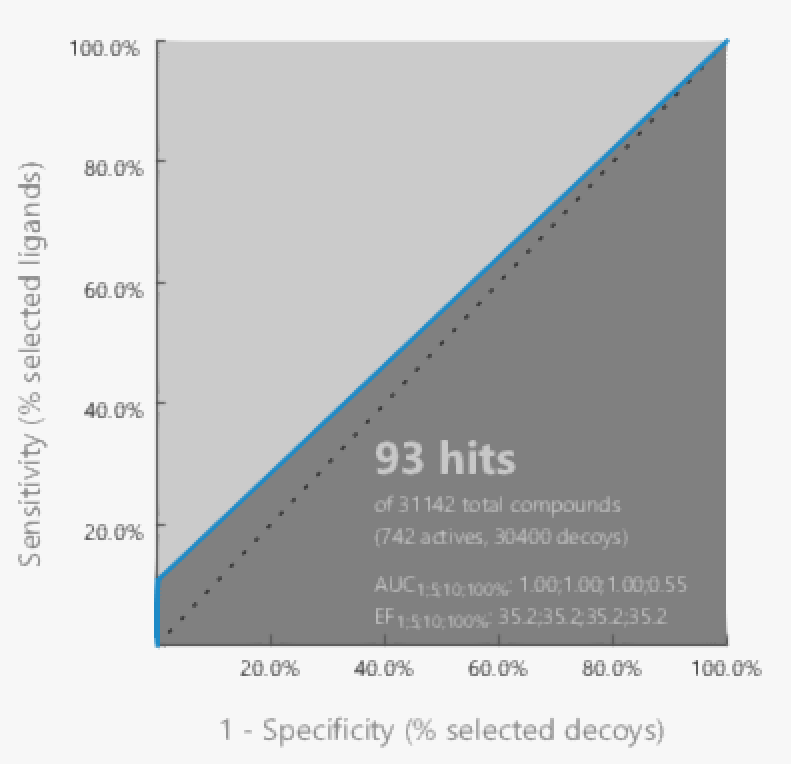
AUC(100%)が0.55となり若干改善されました。
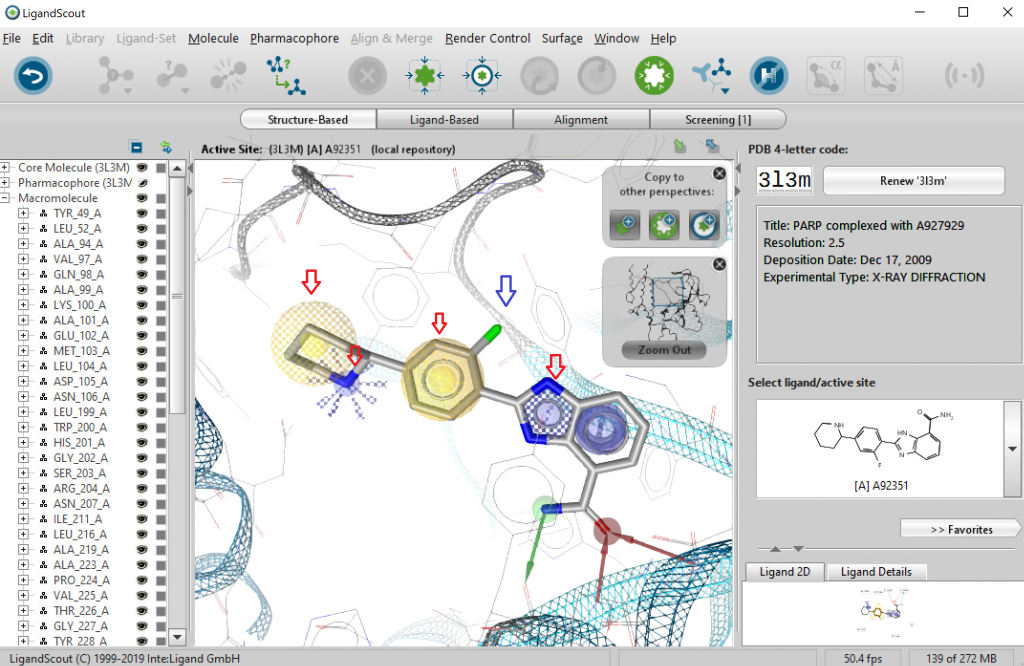
最後にファーマコフォアの修正と設定の変更を行います。赤矢印で示す4つのFeatureをオプションとします([Pharmacophore]→[Mark Features as Optinal])。そして青矢印のFeatureをdeleteキーで削除します。
この操作により、少なくとも3つのFeatureは満たす必要があるが、残りのFeatureに対してはマッチすればスコアに加算されることになります。
PARP1は、NADをADPリボースとニコチンアミドに分解する酵素です。この3つのFeatureはニコチンアミドに相当する部分ですので、オプションにはしませんでした。ここで修正したファーマコフォアを用いて、これまでと同じ手順によりバーチャルスクリーニングを実施します。
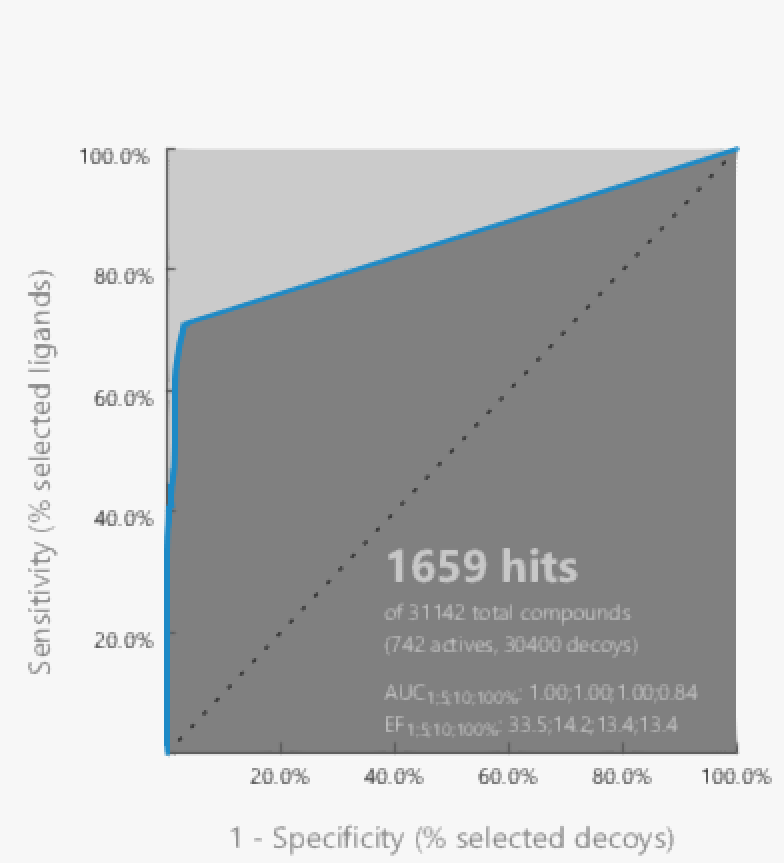
AUC(100%)が0.84、EF(1%)が33.5と大幅に性能が改善されました。
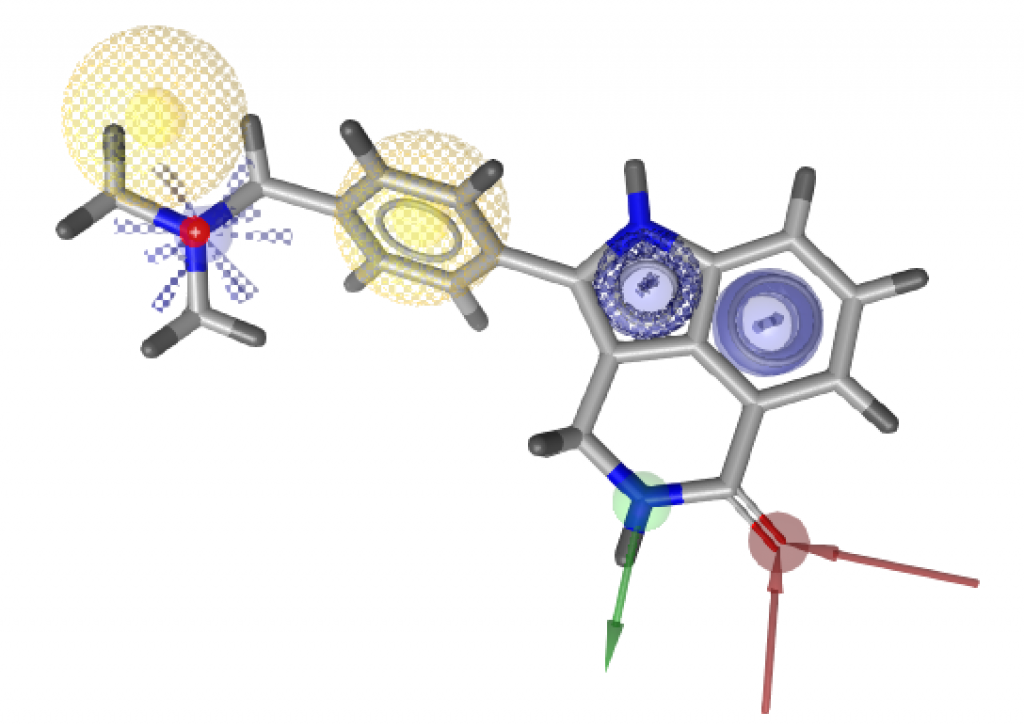
スコアトップの化合物は活性化合物の1つである上図の化合物でした。基質の情報、無視できるFeature数、そしてオプション等を利用することで、単独の結晶構造から構築したファーマコフォアでも良い性能を出すことができます。
Category: LigandScout