今回は弊社のソフトウェア製品であるCzeekSを使って、CGBVS法の予測精度の検証を行いました。
現在弊社で作成済みのCGBVSモデルはChEMBL27のデータを基にしております。ChEMBLdbの最新バージョンは29なので、27から新たに追加された部分についてCGBVSで予測してみました。
検証方法
先ず検証するタンパク質はGPCRとキナーゼにしました。これら2種類のタンパク質は創薬ターゲットとされることが多く、ChEMBLdbに登録されているデータ数も豊富なので機械学習の検証には丁度良いのです。前述したようにChEMBL27のデータで作成したCGBVSモデルを利用して、ChEMBL29のデータにおいてChEMBL27から増えた部分について予測計算をして、予測精度を求めました。ChEMBL29で27から増えているデータは以下の表の通りです。表中の正例は活性値が30uM以下の化合物-タンパク質の組み合わせの数で、負例は活性値が50uM以上の化合物-タンパク質の組み合わせの数です。正例については1万以上データが増えています。
| protein group | ver. 27 正例 | ver. 29 正例 | ver. 27 負例 | ver. 29 負例 |
|---|---|---|---|---|
| GPCR | 358718 | 370323 | 28474 | 28872 |
| キナーゼ | 263319 | 276698 | 12442 | 13101 |
CGBVSモデル(ChEMBL27)
ChEMBL27のデータより構築したGPCRとキナーゼのCGBVSモデルについての情報は以下の通りです。どちらのモデルも5-foldクロスバリデーションが90%以上の良好な予測精度となっています。
GPCRモデル
| 項目 | 値 |
|---|---|
| 化合物数 | 278380 |
| 化合物記述子 | alvaDesc |
| 化合物記述子数 | 941 |
| タンパク質数 | 805 |
| タンパク質記述子 | MSA |
| タンパク質記述子数 | 788 |
| 正例 (<30uM) | 358718 |
| 負例 (>50uM) | 28474 |
| 5-fold CV | 92.66 % |
キナーゼモデル
| 項目 | 値 |
|---|---|
| 化合物数 | 153439 |
| 化合物記述子 | alvaDesc |
| 化合物記述子数 | 941 |
| タンパク質数 | 427 |
| タンパク質記述子 | MSA |
| タンパク質記述子数 | 423 |
| 正例 (<30uM) | 263319 |
| 負例 (>50uM) | 12442 |
| 5-fold CV | 93.76 % |
結果
CGBVSでver.27とver.29の差分についてスコアを計算しました。先ず大まかな予測精度として感度(実験で活性有りとされる化合物を正しく活性有りと予測できた割合)を計算しました。CGBVSのスコアは0~1の範囲の値を取り、この値は対象の化合物が活性を持つ確率値に相当相当します。したがって0.5以上を活性有りと判断することとしています。GPCRについての感度は0.7484で、キナーゼは0.5574でした。GPCRの方はまあまあ良いの精度でしたが、キナーゼについては少々低い値となりました。
各タンパク質毎に結果をまとめたものを以下の表に示します。タンパク質によって予測結果が良かったものと悪かったものと色々とあることが分かります。CGBVSは機械学習を基にした予測モデルなので、
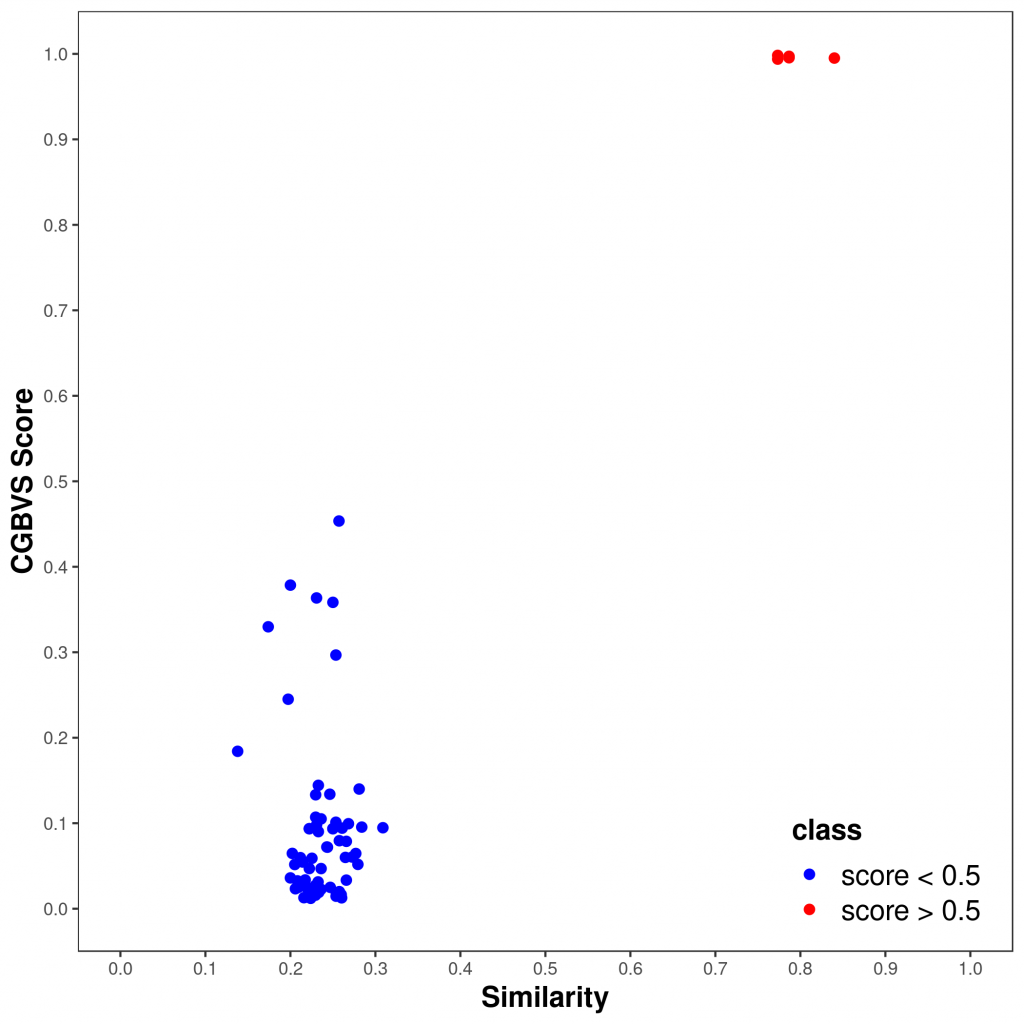
モデル作成に用いた化合物と類似度が小さい化合物の予測が基本的には苦手です。例えばGPCRで予測精度が悪かったCCR2_HUMANが典型的でした。下図は横軸に化合物類似度で縦軸にスコアをとった散布図で、CCR2の計算結果をプロットしています。赤い点はスコアが0.5以上で正しく予測できたもの、
青い点は逆に予測が外れたものです。図を見ると赤い点が右上に偏っています。これは化合物類似度が高いものがスコアが良かったということで、ChEMBL29になって増加したCCR2のデータが、今までの化合物とは類似度が低いものが多いので予測精度が低くなったと言えます。
このような新規構造の化合物の予測が苦手なのは機械学習の宿命ですが、逆に考えるとChEMBL29のデータを学習することにより苦手部分がまた一つ克服できるとも考えられます。我々はこのようにして常に最新のデータを学習させて予測モデルを更新して少しずつ予測精度を高めています。
GPCR
予測精度の良かったもの
| uid | accession | posi27 | diff_posi | TP | specificity |
|---|---|---|---|---|---|
| CXCR3_HUMAN | P49682 | 1331 | 163 | 163 | 1.000000 |
| FFAR1_HUMAN | O14842 | 1375 | 110 | 110 | 1.000000 |
| GLR_HUMAN | P47871 | 1004 | 49 | 49 | 1.000000 |
| S1PR1_HUMAN | P21453 | 2537 | 33 | 33 | 1.000000 |
| S1PR3_HUMAN | Q99500 | 1053 | 27 | 27 | 1.000000 |
| CRFR1_HUMAN | P34998 | 1850 | 20 | 20 | 1.000000 |
| MCHR1_HUMAN | Q99705 | 3627 | 15 | 15 | 1.000000 |
| GPR88_HUMAN | Q9GZN0 | 203 | 12 | 12 | 1.000000 |
| S1PR2_HUMAN | O95136 | 259 | 12 | 12 | 1.000000 |
| GIPR_HUMAN | P48546 | 119 | 8 | 8 | 1.000000 |
予測精度の悪かったもの
| uid | accession | posi27 | diff_posi | TP | specificity |
|---|---|---|---|---|---|
| GASR_HUMAN | P32239 | 1411 | 51 | 2 | 0.039216 |
| CCR2_HUMAN | P41597 | 1732 | 73 | 5 | 0.068493 |
| CCKAR_HUMAN | P32238 | 597 | 55 | 4 | 0.072727 |
| APJ_HUMAN | P35414 | 136 | 107 | 8 | 0.074766 |
| O51E2_HUMAN | Q9H255 | 0 | 24 | 2 | 0.083333 |
| BKRB2_HUMAN | P30411 | 491 | 12 | 1 | 0.083333 |
| NPY5R_HUMAN | Q15761 | 1115 | 10 | 1 | 0.100000 |
| ADA2A_HUMAN | P08913 | 1060 | 57 | 7 | 0.122807 |
| SSR2_HUMAN | P30874 | 622 | 22 | 3 | 0.136364 |
| HRH2_HUMAN | P25021 | 404 | 49 | 7 | 0.142857 |
キナーゼ
予測精度の良かったもの
| uid | accession | posi27 | diff_posi | TP | specificity |
|---|---|---|---|---|---|
| PMYT1_HUMAN | Q99640 | 25 | 9 | 9 | 1.000000 |
| KS6B2_HUMAN | Q9UBS0 | 26 | 1 | 1 | 1.000000 |
| PKN3_HUMAN | Q6P5Z2 | 4 | 1 | 1 | 1.000000 |
| TSSK6_HUMAN | Q9BXA6 | 0 | 1 | 1 | 1.000000 |
| WNK1_HUMAN | Q9H4A3 | 12 | 1 | 1 | 1.000000 |
| WNK2_HUMAN | Q9Y3S1 | 3 | 1 | 1 | 1.000000 |
| MTOR_HUMAN | P42345 | 7155 | 127 | 124 | 0.976378 |
| PLK1_HUMAN | P53350 | 21817 | 72 | 69 | 0.958333 |
| MK01_HUMAN | P28482 | 12252 | 71 | 67 | 0.943662 |
| MK03_HUMAN | P27361 | 231 | 43 | 40 | 0.930233 |
予測精度の悪かったもの
| uid | accession | posi27 | diff_posi | TP | specificity |
|---|---|---|---|---|---|
| IKKE_HUMAN | Q14164 | 641 | 44 | 1 | 0.022727 |
| KPCD1_HUMAN | Q15139 | 324 | 31 | 1 | 0.032258 |
| RIPK2_HUMAN | O43353 | 149 | 55 | 2 | 0.036364 |
| TBK1_HUMAN | Q9UHD2 | 693 | 54 | 2 | 0.037037 |
| ERN1_HUMAN | O75460 | 460 | 26 | 1 | 0.038462 |
| GRK5_HUMAN | P34947 | 209 | 24 | 1 | 0.041667 |
| KS6A6_HUMAN | Q9UK32 | 56 | 24 | 1 | 0.041667 |
| PKN1_HUMAN | Q16512 | 66 | 23 | 1 | 0.043478 |
| KPCI_HUMAN | P41743 | 804 | 36 | 2 | 0.055556 |
| AAPK1_HUMAN | Q13131 | 612 | 124 | 9 | 0.072581 |
Category: CGBVS/CzeekS, Machine Learning