今回は、PARP-1のファーマコフォアを使って、Ligand Expoに登録されている化合物データに対して探索を行ってみます。
Step 1: Ligand Expoから化合物データの取得
ここから以下の2つのファイルを取得します。
Components-pub.sdf.gz:
PDBに登録されている低分子化合物のIDと構造情報
cc-to-pdb.tdd:
低分子化合物のIDとPDB IDの関係を示した情報
Components-pub.sdf.gzは解凍して前回の記事の方法(step 3)で配座生成を行います。
Step 2: PARP-1のファーマコフォアデータの保存と読み込み
前回の記事の方法(step 2)で作成したファーマコフォアにExclusion Volumes Coatを追加したファーマコフォアをPML形式で保存します。
Exclusion Volumes Coatの追加方法:
Structure-Based Perspecitveにおいて[Pharmacophore]→[Add Exclusion Volumes Coad]
Pharmacophoreの保存方法:
Structure-Based Perspectiveにおいて[File]→[Save as File]でpmz形式で保存する。ここではPARP1.pmzで保存します。
Pharmacophoreの読み込み方法:
Screening Perspectiveにおいて[File]→[Open]でPARP1.pmzを読み込みます。
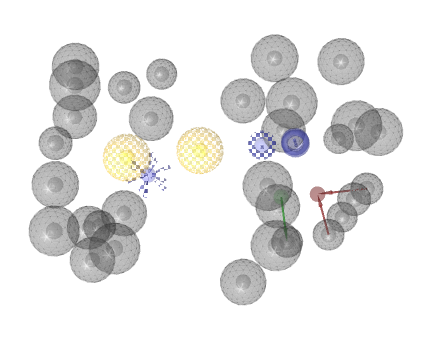
Step 3: Ligand Expoに対するファーマコフォア探索
Screening PerspectiveにおいてComponents-pub.ldbを読み込んでファーマコフォア探索を実施します。Components-pub.ldbには約28,000の化合物が登録されています。
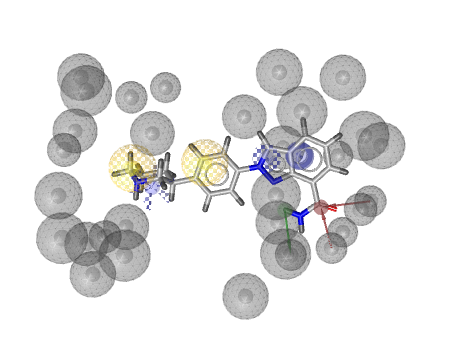
上図は、Relatieve Pharmacophore-Fit Scoreの最も良い化合物となります。
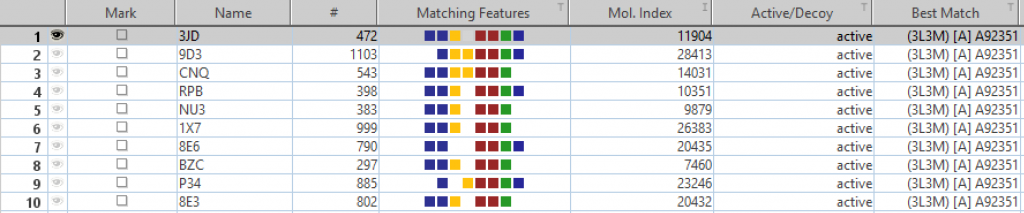
上位10化合物のうち6化合物がPARP-1の阻害剤でした。
今回利用したファーマコフォアは、なかなか選択性の高いものだと思います。
一方で、PARP-1阻害剤ではない上位の化合物は、擬陽性だと思われますが、PARP-1を阻害する可能性もあると思います。ドラッグリポジショニングの用途でも利用できるかもしれません。
Name列のIDが低分子化合物のIDとなります。PDBとの関係は、
cc-to-pdb.tddに記載されています。
Category: LigandScout